2021.06.04.32
Files > Volume 6 > Vol 6 No 4 2021
Pruebas moleculares para el diagnóstico de COVID-19: La respuesta de Sudamérica
Molecular tests for the diagnosis of COVID-19: South America's response

Pool Marcos-Carbajal1,2,, Christian Allca-Muñoz14,, Macarena Ganoza-Farro1,4,, André Valdez-Olivera1,4,, Allison Gomez-Martel1,4, , Maribel Huaringa-Nuñez5,, Alberto Salazar-Granara1,3,
Available from: http://dx.doi.org/10.21931/RB/2021.06.04.32
RESUMEN
Actualmente a nivel mundial se utiliza la reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR) como una técnica de alta precisión en la detección y amplificación de secciones específicas de la estructura génica. En tal sentido es considerado como prueba diagnóstica molecular en la detección del SARS-CoV-2. Se realizó una revisión de la literatura en artículos publicados en los últimos 5 años (2015 – 2020). Se consultaron las bases de datos: PUBMED (Medline), PUBMED CENTRAL, y SCIELO. Se recuperaron artículos en español, portugués, e inglés, mediante los términos de búsqueda SARS-CoV-2, RT-PCR, y COVID-19. Las RT-PCR que utilicen especímenes de hisopado nasal y faríngeos son considerados como gold standard en casos sospechosos de COVID-19. Mediante lo revisado a nivel de Sudamérica, la RT-PCR resultó ser la prueba de elección durante el período menor a 7 días de infección, resaltando la producción in house en escala por Uruguay y la Prueba de amplificación isotérmica mediada por bucle (RT-LAMP) adaptado en Perú, como una alternativa diagnóstica rápida con principios similares a la RT-PCR, solo que para establecimientos de salud con menor equipamiento, e infraestructura y personal entrenado. La disponibilidad de pruebas diagnósticas moleculares también es crucial para el aislamiento de casos positivos y el seguimiento de la cadena epidemiológica de transmisión.
Palabras clave. COVID-19; Virus SARS; Diagnóstico; RT-PCR (Fuente: DeCS BIREME).
Palabras clave. COVID-19; Virus SARS; Diagnóstico; RT-PCR (Fuente: DeCS BIREME).
ABSTRACT
Currently, worldwide, the reverse transcriptase-polymerase chain reaction (RT-PCR) is used as a high-precision technique in detecting and amplifying specific sections of the genetic structure. In this sense, it is considered a molecular diagnostic test in the detection of SARS-CoV-2. A review of the literature was carried out on articles published in the last 5 years (2015-2020). The following databases were consulted: PUBMED (Medline), PUBMED CENTRAL, and SCIELO. Articles in Spanish and English were retrieved, according to MESH terminology: SARS-CoV-2, RT-PCR, COVID-19. RT-PCR tests that use nasal and pharyngeal swab samples are considered the standard gold test in suspected cases of COVID-19. Through the South American review of molecular tests, RT-PCR turned out to be the test of choice in the countries of South America during the period less than 7 days of infection, highlighting the in-house production in scale by Uruguay and the Loop-mediated isothermal amplification test (RT-LAMP) adapted in Peru as a rapid diagnostic alternative with principles similar to RT-PCR, only for health facilities with less equipment, infrastructure, and trained personnel. The availability of molecular diagnostic tests is also crucial for isolating positive cases and monitoring the epidemiological chain of transmission.
Keywords. COVID-19; Virus SARS; Diagnosis; RT-PCR (Source: DeCS BIREME).
INTRODUCCIÓN
Los coronavirus son un grupo de virus ARN altamente diversos de la familia Coronaviridae que se dividen en 4 géneros: alfa, beta, gamma y delta, y que causan enfermedades de leves a graves en humanos y animales 1. Sin embargo, tres coronavirus zoonóticos que causan enfermedades graves en humanos han emergido: el coronavirus del síndrome respiratorio agudo grave (SARS-CoV) en 2002-2003, el coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV) 2; y, con un impacto aún mayor, el síndrome respiratorio agudo severo tipo-2 (SARS-CoV-2) 3. En enero de 2020, el agente etiológico responsable de un grupo de casos de neumonía grave en Wuhan, China, fue identificado como un nuevo betacoronavirus inicialmente llamado 2019-nCoV, actualmente SARS-CoV-2, distinto del SARS-CoV y MERS-CoV 4. La secuencia genómica completa de este nuevo agente está disponible y se han desarrollado diferentes protocolos y técnicas para su detección, aunque aún no se han validado por completo 5. La introducción de un caso sospechoso relacionado con el 2019-nCoV en la Región de las Américas colocó en alerta a la Organización Panamericana de la Salud/Organización Mundial de la Salud (OPS/OMS) recomendando a los Estados Miembros garantizar su identificación oportuna, el envío de las muestras a laboratorios nacionales o de referencia y la implementación del protocolo de detección molecular para 2019-nCoV 6,7. La OMS puso por su parte a disposición dos protocolos de diagnóstico molecular en la página web “Pruebas de laboratorio para casos humanos sospechosos del nuevo coronavirus 2019 (2019-nCoV)” 8. Por tanto, hay una necesidad inmediata de un procedimiento de prueba de detección de primera mano, que debe estar disponible para personas sintomáticas y asintomáticas que tengan contacto estrecho con un individuo infectado 9. De esta manera se puede determinar si la persona probada es negativa o potencialmente positiva para la presencia de SARS-CoV-2. Las plataformas de detección moleculares disponibles actualmente para el SARS-CoV-2, se basan en un concepto simple de detección indirecta de ARN por una prueba de reacción de cadena de polimerasa con transcripción inversa (RT-PCR) y la posterior clonación y uso de ADN complementario (ADNc) en otras técnicas moleculares. En resumen, una persona portadora potencial es sometido a una colección de frotis faríngeo (frotis de garganta) y/o esputo, esta muestra recolectada luego se procesa a través de una serie de procedimientos para purificar ARN viral, convertir ARN en ADNc mediante una transcriptasa inversa y medición del número de copias mediante gPCR utilizando conjuntos de cebadores específicos obtenidas de la secuencia genómica de SARS-CoV-2 10,11. Es así que, utilizando estos recursos disponibles, es posible que se pueda probar la presencia de SARS-CoV-2 de forma segura en un laboratorio certificado para Bioseguridad de Nivel II, mínimamente equipado con una máquina gPCR 12. Con el fin de desarrollar una plataforma molecular rápida, de alta precisión, de bajo costo, lista para el laboratorio y protocolos de laboratorio seguro para evaluar sujetos asintomáticos como un procedimiento de detección de primera mano para la detección del SARS-CoV-2.
El objetivo de este estudio fue identificar todos los recursos disponibles que se han desarrollado en los países de Sudamérica para el diagnóstico molecular de SARS-CoV-2
VIROLOGÍA DEL CORONAVIRUS
El genoma del SARS-CoV-2 contiene elementos específicos que facilitan la replicación del virus y la formación de la nucleocápside y la proteína S de anclaje. El genoma está compuesto de segmentos estructurales y no estructurales que son utilizados como dianas en pruebas moleculares mostrada en la Figura 1.
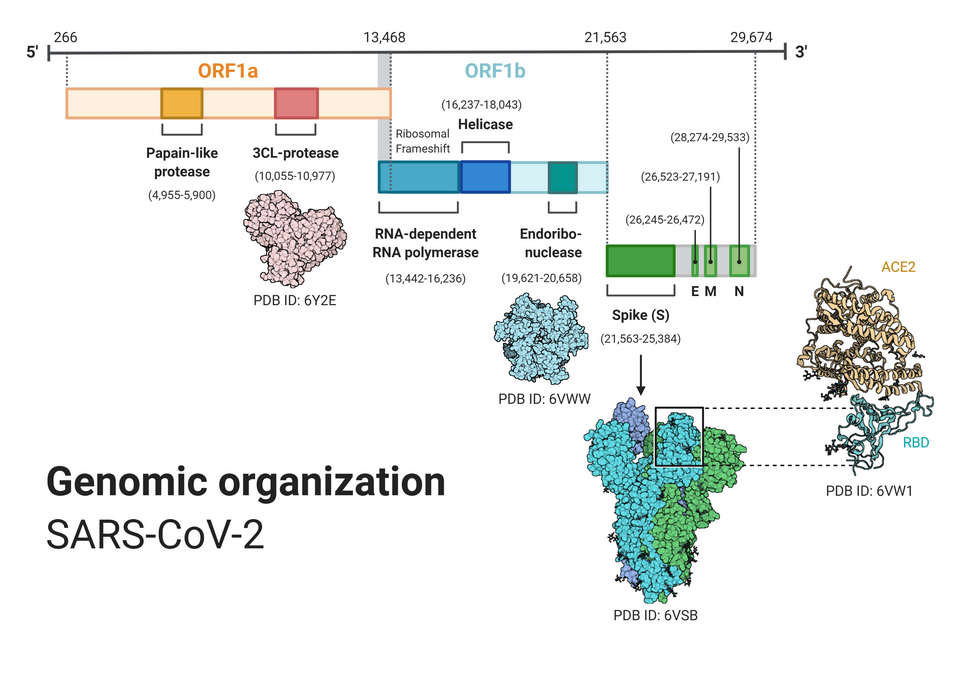
Figura 1. Estructura genómica del SARS-CoV-2
PRUEBAS MOLECULARES SARS-CoV-2 EN EL MUNDO
El examen molecular que se está empleando en todo el mundo para la detección directa de la infección con el SARS-CoV-2 es la prueba de gPCR sobre los genes expresados por este virus. Se realizó el secuenciamiento del fragmento génico ORF1ab y de un fragmento de la proteína de la nucleocápside (NP). Para un diagnóstico confirmatorio de SARS-CoV-2, la OMS indica la detección del gen de la proteína E, seguida de la expresión del gen RdRp y la expresión del gen N solo si se requiriese un ensayo confirmatorio adicional 13,14.
Wang, et al. analizaron diferentes muestras: nasales, hisopados faríngeos, sangre, esputo, heces y orina provenientes de pacientes infectados con el virus en 3 hospitales de China. En pacientes graves en ventilación mecánica se recolectaron muestras del líquido de lavado broncoalveolar y de biopsia de cepillo de fibrobroncoscopio, para ser utilizado como fuente para la posterior extracción de material genético (ARN), RT-PCR y diagnóstico. Se realizó la técnica de reacción de cadena de polimerasa con transcripción inversa (RT-PCR) en la cual se determinó dos genes dianas del fragmento génico para la detección de SARS-CoV-2. El secuenciamiento incluyó el marco de lectura abierto 1ab (ORF1ab) y fragmento de proteína de la nucleocápside (NP). Se tomaron 1070 pruebas provenientes de 205 pacientes infectados por SARS-CoV-2, en la cual el lavado broncoalveolar demostró tener un 93% de positividad para diagnóstico de SARS-CoV-2, seguido por esputo, 72%; hisopado nasal, 63%; biopsia con cepillo de fibrobroncoscopia, 46%; hisopado faríngeo, 32%; heces, 29% y sangre, 1%; mientras que ninguna prueba con muestra de orina resultó positiva 15, 16.
Bajo la misma técnica, Liu et al. estudiaron una población de 4880 personas, las cuales 1875 fueron positivas para SARS-CoV-2, determinando diferentes tipos de detección dependiendo a qué gen diana iba dirigido. Para el gen ORF 1ab fueron positivos 40.98% y para el fragmento de proteína de la nucleocápside (NP) fueron positivos 39.80%. Siendo la muestra de lavado broncoalveolar de mayor positividad, 80% para NP, 100% para ORF1ab y 80% para ambos genes; seguido de esputo 49,12% para NP, 50.88% para ORF1ab y 49.12% para ambos genes; y hisopado nasal y faríngeo 39.64% para NP, 40.81% para ORF1ab y 38.25% para ambos genes 11.
Adicionalmente, la utilización de primers para la detección de SARS-CoV-2 para poder diferenciar de otros SARS-CoV fue propuesta por Corman et al. En las cuales se diseñan fragmentos de secuenciamiento genético para ORF1ab RdRp para la posición 15361-15460 nts; para el fragmento de la proteína N en la posición 28555-28682 nts; y para el gen de la proteína E en la posición 26141-26253 nts. Para la muestra obtenida proveniente de Wuhan en el 2019 (NM908947 Wuhan-Hu-1) comparadas con otras muestras virales (NC-004718 Sars-CoV) y según los primers diseñados se logró demostrar que todos eran una especie de SARS-CoV y de la subfamilia betacoronavirus. Ante ello se propuso analizar con otras muestras virales de la misma subfamilia, entre ellas, muestras que provenían de la ciudad de Wuhan como, BetaCoV/Wuhan/PBCAMS-WH-01/2019|EPI_ISL_402123; BetaCoV/Wuhan/IVDC-HB-01/2019|EPI_ISL_402119; BetaCoV/Wuhan/IVDC-HB-04/2020|EPI_ISL_402120; BetaCoV/Wuhan/IVDC-HB-05/2020|EPI_ISL_402121 y BetaCoV/Wuhan/WIV04/2019|EPI_ISL_402124; y muestras virales provenientes de murciélagos, MG772933 Bat SARSrelated CoV (bat-SL-CoVZC45) y NC_014470 Bat SARS-related CoV (BM48-31/BGR/2008). Demostrando variación en la secuencia de nucleótidos para los genes RdRp y la proteína de la nucleocápside, pero sin encontrar diferencia para los genes de la proteína E, por lo cual se concluye la variabilidad con respecto a la secuencia de nucleótidos entre la misma familia de SARS y su importancia para el diseño y estandarización de primers para la detección específica de SARS-CoV-2 para su diagnóstico 10.
Sin embargo, el Centro para el Control y la Prevención de Enfermedades (CDC por sus siglas en inglés) en los Estados Unidos utiliza un método de detección diferente a lo establecido por la OMS. Esta está enfocada en la detección principal del gen N1, N2, N3 y RP dando resultado positivo con valores de < 40.00 Ct 17.
METODOLOGÍA
El estudio presenta un registro nacional de investigación en salud con código EI00001613. Es de tipo observacional, descriptivo, transversal y retrospectivo. La revisión sistemática del presente estudio abarca criterios para la inclusión de las diferentes pruebas moleculares realizadas en Latinoamérica para la detección del SARS-CoV-2.
Criterios de elegibilidad
Se cuenta con revisión bibliográfica internacional de artículos publicados en las bases de datos en línea PUBMED (https://pubmed.ncbi.nlm.nih.gov/) y SCIELO (https://scielo.org/), seleccionando artículos de los últimos 5 años (2015-2020). Se utilizaron los siguientes términos de búsqueda según terminología DeCS BIREME: COVID-19; Virus SARS; Diagnosis; RT-PCR. Se incluyeron estudios en idiomas de inglés, portugués y español.
Estrategia de búsqueda
Se realizaron las siguientes búsquedas: (COVID-19 OR SARS-Cov-2 OR Coronavirus OR COVID-19 Testing) AND (RT-PCR OR Diagnosis OR Polymerase Chain Reaction OR COVID-19 Nucleic Acid Testing OR Real-Time Polymerase Chain Reaction OR Reverse Transcriptase Polymerase Chain Reaction OR Multiplex Polymerase Chain Reaction OR PCR). Con filtros de búsqueda para: Abstract, Free full text, Classical Article, Review, in the last 5 years, Humans, English, Spanish, Adult: 19+ years. Las búsquedas se complementaron con revisiones específicas de las siguientes páginas web: Organización Mundial de la Salud (https://www.who.int/es), Agencia Nacional de Vigilancia de Salud-Brasil (https://www.gov.br/anvisa/pt-br), Federación Médica de la Provincia de Buenos Aires-Argentina (http://www.femeba.org.ar) y Ministerio de Salud-Perú (https://www.gob.pe/minsa/). Ministerio de Salud de Bolivia (https://www.minsalud.gob.bo/).
PRUEBAS MOLECULARES EN SUDAMÉRICA
Las situaciones variadas de cada país en sudamérica los ha llevado a la utilización de pruebas diagnósticas como parte de un plan estratégico complementario para el SARS-CoV-2 en caso de que los reactivos para la prueba de RT-PCR no se puedan sustentar durante el tiempo.
Para realizar las diferentes pruebas de detección de SARS-CoV-2 deben primero tener la presencia de criterios clínicos, como la afectación de vía respiratoria asociada a fiebre, o sensación febril, tos, secreción nasal, dolor de garganta o dificultad para respirar. En aquellos casos se realizará la prueba de RT-PCR para detección de SARS-CoV-2, influenza y presencia de virus sincitial respiratorio 18.
En Brasil se utilizan diferentes pruebas de diagnóstico aprobados en un informe de la Agencia Nacional de Vigilancia Sanitaria (ANVISA) en la fecha del 12 de abril del 2020, en la cual se detallan diferentes pruebas recolectadas en base de datos de ANVISA (19) (Tabla 1).

Tabla 1. Métodos Diagnósticos para SARS-CoV-2 en BRASIL
En un informe aprobado el día 21 de abril del 2020 en Argentina por la Administración Nacional de Medicamentos, Alimentos y Tecnología Médica (ANMAT) se utiliza como método de diagnóstico la técnica de RT-PCR. Los genes a los cuales apuntan la detección son; N, E, S, y RdRp, utilizando gabinetes de bioseguridad en instalaciones BSL-2 o su equivalente 20.
En Uruguay se ha planteado la utilización de una prueba RT-PCR Real TM Fast (HEX) desarrollada in house para la detección y diagnóstico de SARS-CoV-2. El principio de la técnica se basa en la detección del gen que codifica la proteína N, gen humano de la RNasa P (control humano interno), pudiendo utilizar dos tipos de termocicladores, ROTOR GENE y ABI 7500. Dando una muestra positiva ct <35 con un control positivo de <35 21.
En Perú a mediados de septiembre se empezó a utilizar la prueba molecular rápida del tipo LAMP (amplificación isomérica mediada en lazo), la cual disminuye el tiempo de espera para el diagnóstico de SARS-CoV-2, la cual lleva un resultado en dos horas. Esta prueba de PCR-LAMP se enfoca en la detección también del gen N, ORF 1ab y S, demostrando una sensibilidad entre 80 y 100%, y una especificidad entre 73 y 100% para el diagnóstico de SARS-CoV-2 22.
Países como Chile, Ecuador y Colombia en la detección del SARS-CoV-2 emplean la técnica de RT-PCR elaborada por Corman y colaboradores en Berlín, la cual se basa en la detección del gen de la proteína E principalmente para luego dar una confirmación de un segundo ensayo para el gen RdRp (10). Los diferentes ensayos establecidos en cada país se detallan en la tabla 2.
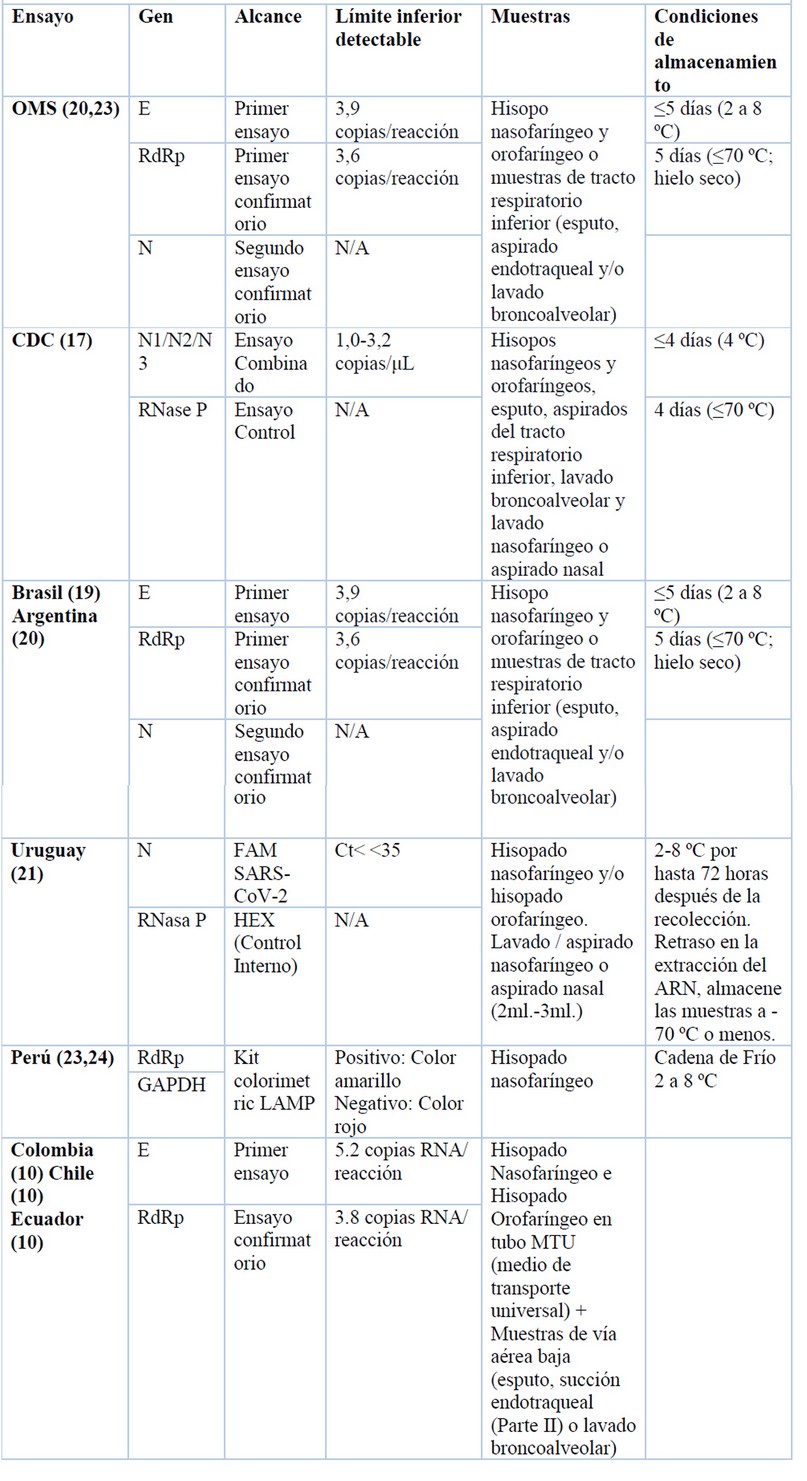
Tabla 2. Comparación de pruebas moleculares para diagnósticos para SARS-CoV-2
En Bolivia las pruebas de diagnóstico han sido manejadas con métodos moleculares, entre ellos la prueba de PCR que detecta 2 marcadores del genoma del virus SARS-Cov-2 el gen E y gen RdRP. 25,26 En pruebas rápidas moleculares el país adquirió xpert Xpress SARS-CoV-2 test (cepheid) y el abbott ID NOW COVID-19 test (Abbott). 27,28
En Trinidad y Tobago las pruebas de mayor alcance y uso han sido las pruebas de antígeno para las cuales se piden muestra tipo nasal y nasofaríngeo, y las pruebas de amplificación de ácido nucleico para el SARS-CoV-2 que identifican específicamente las secuencias de ARN con muestras de tipo nasal, nasofaríngeo, orofaríngeo, esputo y saliva. 29,30 Sin embargo, se han implementado métodos diferentes para amplificar los ácidos nucleicos y detectar el virus que incluye reacción de amplificación de endonucleasa de mellado (NEAR), amplificación mediada por transcripción (TMA), amplificación isotérmica mediada por bucle (LAMP), amplificación dependiente de helicasa (HDA), repeticiones palindrómicas cortas agrupadas regularmente interespaciales (CRISPR) y amplificación de desplazamiento de hebra (SDA). 31
En Paraguay en asociación con la OPS se estableció como técnica estándar el uso de la prueba de Reacción en Cadena de la Polimerasa en tiempo real (RT-PCR) para la detección del SARS-CoV-2 con muestras de hisopado nasal y faríngeo recolectadas mediante técnica estándar en medio de cultivo viral y transportadas al centro de referencia, en cadena de frío. Siguiendo las recomendaciones de la OPS, Paraguay optó por la detección de dos genes: el gen E (cuya presencia indica que pertenece a la familia de beta coronavirus) y RdRp (cuya presencia es específica para el SARS-CoV-2 y sirve como confirmación). 32, 33
CONCLUSIONES
Las pruebas moleculares están basadas en la técnica de transcripción reversa seguida de la reacción en cadena de polimerasa cuantitativa (qRT-PCR) denominada como el Gold estándar para detección de SARS-CoV-2. Esta prueba detecta genes conservados del virus como el gen RNA dependiente de RNA polimerasa (RdRp), el gen de la cápside (E) y el gen de la proteína Spike (S); actualmente existen kits moleculares en los que se identifica más de un gen, mejorando así la especificidad y eficiencia de la prueba. Para la detección de genes virales se usan muestras de hisopado nasofaríngeo, esputo y lavado bronquio-alveolar de una persona infectada, siendo estos dos últimos los de mejor sensibilidad; esta técnica, en algunos casos, permite la detección incluso hasta una semana antes del inicio de los síntomas. Además del qRT-PCR, existe otra técnica molecular siendo implementada recientemente para la detección de SARS-CoV-2, la transcripción reversa seguida de una amplificación isotérmica mediada por lazo (RT– LAMP) desarrollada por Baek y colaboradores a inicios de este año. En este ensayo se utilizan primers diseñados para amplificar genes del virus SARS-CoV-2, como por ejemplo el gen de la nucleocápside del RNA viral, como es mostrado en otros estudios, los genes ORF1ab y Spike. RT-LAMP genera resultados en aproximadamente 30 minutos y es interpretado por una evaluación colorimétrica que no tiene reacción cruzada aparente con otros coronavirus, virus de influenza u otros virus respiratorios. Tanto qRT-PCR como RT-LAMP tienen alta especificidad en casos de bajo o nulo sesgo operacional, sin embargo, la existencia de un error en la toma de muestra puede dar como resultado un falso negativo siendo este uno de los problemas más frecuentes. Eventualmente la falta de experiencia del personal de colecta, dan como resultado una amplificación insatisfactoria o un resultado falso negativo. Un paciente falso negativo es un riesgo para la salud pública por la capacidad de diseminación, obstaculizando así el objetivo de contener la propagación del virus.
AGRADECIMIENTOS
Los autores agradecen al Centro de Investigación en Medicina Tradicional y Farmacología (CIMTFAR) de la Facultad de medicina Humana Universidad San Martin de Porres (USMP) y al Laboratorio de Investigación en Biología Molecular (LIBM) de la Escuela Profesional de la Universidad Peruana Unión UPeU) y. A las autoridades de ambas instituciones Dr. Frank Lizaraso Caparó y Dr. Luis Felipe Segura Chavez por el apoyo y licencias para el desarrollo de la investigación.
CONTRIBUCIONES DE AUTORÍA
P. Marcos ha participado en la concepción y diseño del artículo. Los procedimientos y resultados fueron realizados por P. Marcos, C. Allca y A. Salazar. Los análisis y discusiones fueron realizados por P. Marcos, C. Allca, M. Ganoza, A. Valdez, M. Huaringa y A. Salazar. La redacción del artículo estuvo a cargo de P. Marcos, C. Allca, A. Valdez, M. Ganoza, M. Huaringa y A. Salazar. La revisión crítica la realizó A. Salazar. La versión final estuvo a cargo de todos los autores.
FUENTES DE FINANCIAMIENTO
El artículo fue autofinanciado por la Universidad de San Martín de Porres y por la Universidad Peruana Unión.
CONFLICTO DE INTERÉS.
Los autores declaran no tener conflicto de interés.
Los autores declaran no tener conflicto de interés.
DECLARACIÓN DE ÉTICA
Los autores confirman que están de acuerdo con las políticas éticas de la revista. No se requirió aprobación ética ya que esta es un artículo de revisión sin datos de investigación originales.
REFERENCIAS
- Hui DSC, Zumla A. Severe Acute Respiratory Syndrome. Infect Dis Clin North Am. diciembre de 2019;33(4):869-89.
- de Wit E, van Doremalen N, Falzarano D, Munster VJ. SARS and MERS: recent insights into emerging coronaviruses. Nat Rev Microbiol. 2016;14(8):523-34.
- V'kovski P, Kratzel A, Steiner S, Stalder H, Thiel V. Coronavirus biology and replication: implications for SARS-CoV-2. Nat Rev Microbiol. 28 de octubre de 2020;1-16.
- World Health Organization. Laboratory testing of human suspected cases of novel coronavirus (nCoV) infection - Interim guidance. WHO/2019-nCoV/laboratory/2020.1. [Online] January 17, 2020.https://www.who.int/health-topics/coronavirus/laboratory-diagnostics-for-novel-coronavirus.
- Instituto de Salud Carlos III. ESTRATEGIA DE DETECCIÓN PRECOZ, VIGILANCIA Y CONTROL DE COVID-19 [Internet]. Ministerio de Sanidad. Gobierno de España; 202, p. 1-32. Disponible en: https://www.mscbs.gob.es/profesionales/saludPublica/ccayes/alertasActual/nCov/documentos/COVID19_Estrategia_vigilancia_y_control_e_indicadores.pdf
- Cronología de la respuesta de la OMS a la COVID-19 [Internet]. [citado 15 de mayo de 2021]. Disponible en: https://www.who.int/es/news/item/29-06-2020-covidtimeline
- Villegoureix I. OPS/OMS | 5 de febrero de 2020: Nuevo coronavirus (nCoV) - Actualización Epidemiológica [Internet]. Pan American Health Organization / World Health Organization. 2020 [citado 15 de mayo de 2021]. Disponible en: https://www3.paho.org/hq/index.php?option=com_content&view=article&id=15709:3-february-2020-novel-coronavirus-ncov-epidemiological-update&Itemid=42346&lang=es
- Detección diagnóstica de coronavirus Wuhan 2019 por RT-PCR en tiempo real - Charité, Berlín-Alemania
- Información básica sobre la COVID-19 [Internet]. [citado 15 de mayo de 2021]. Disponible en: https://www.who.int/es/news-room/q-a-detail/coronavirus-disease-covid-19
- Corman VM, Landt O, Kaiser M, Molenkamp R, Meijer A, Chu DKW, Bleicker T, Brünink S, Schneider J, Schmidt ML, Mulders DGJC, Haagmans BL, van der Veer B, van den Brink S, Wijsman L, Goderski G, Romette JL, Ellis J, Zambon M, Peiris M, Goossens H, Reusken C, Koopmans MPG, Drosten C (2020) Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. doi: 10.2807/1560-7917. ES.2020.25.3.2000045.
- Liu R, Han H, Liu F, Lv Z, Wu K, Liu Y, et al. Positive rate of RT-PCR detection of SARS-CoV-2 infection in 4880 cases from one hospital in Wuhan, China, from Jan to Feb 2020. Clin Chim Acta Int J Clin Chem. junio de 2020;505:172-5.
- Centers for Disease Control and Prevention (2020) Interim laboratory biosafety guidelines for handling and processing specimens associated with Coronavirus disease 2019 (COVID-19) [Internet]. Centers for Disease Control and Prevention, Atlanta. Available from: https://www.cdc.gov/coronavirus/2019-ncov/lab/lab-biosafety-guidelines.html.
- Aguilar Ramírez Priscilia, Enriquez Valencia Yanina, Quiroz Carrillo Carlos, Valencia Ayala Edward, de León Delgado Joel, Pareja Cruz Arturo. Pruebas diagnósticas para la COVID-19: la importancia del antes y el después. Horiz. Med. [Internet]. 2020 Abr [citado 2021 Mayo 12] ; 20( 2 ): e1231. Disponible en: http://www.scielo.org.pe/scielo.php?script=sci_arttext&pid=S1727-558X2020000200014&lng=es. http://dx.doi.org/10.24265/horizmed.2020.v20n2.14.
- Lippi G, Simundic AM, Plebani M. Potential preanalytical and analytical vulnerabilities in the laboratory diagnosis of coronavirus disease 2019 (COVID-19). Clin Chem Lab Med. 2020.
- Wang W, Xu Y, Gao R, Lu R, Han K, Wu G, et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA [Internet]. 11 de marzo de 2020 [citado 15 de mayo de 2021]; Disponible en: https://jamanetwork.com/journals/jama/fullarticle/2762997
- Wang D, Hu B, Hu C, Zhu F, Liu X, Zhang J, et al. Clinical Characteristics of 138 Hospitalized Patients With 2019 Novel Coronavirus–Infected Pneumonia in Wuhan, China. JAMA. 17 de marzo de 2020;323(11):1061.
- Centers for Disease Control and Prevention. CDC 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Diagnostic Panel. https://www.fda.gov/media/134922/download. Last accessed, March 9, 2020.
- Agência Nacional de Vigilância Sanitária - Anvisa [Internet]. [citado 15 de mayo de 2021]. Disponible en: https://www.gov.br/anvisa/pt-br/pagina-inicial
- Ministério da Saúde, Brasil. Brasil - PATRIAVACINADA [Internet]. [citado 15 de mayo de 2021]. Disponible en: https://www.gov.br/saude/pt-br/vacinacao/#diagnostico
- Red Argentina Pública de Evaluación de Tecnologías Sanitarias. Diferentes tipos de tests y estrategias diagnósticas en el contexto de pandemia por COVID-19. 22 de abril de 2020;1-53.
- Abin A. COVID-19 RT-PCR Real TM FAST (HEX). Universidad de la República de Uruguay. Version 2AT Gen. IDK 71. 5 de mayo de 2020;4.
- Adolfo Aramburu; Fabiola Huaroto; Nora Reyes. Precisión diagnóstica de pruebas basadas en amplificación isotérmica mediada en lazo de transcriptasa inversa (RT-LAMP) para SARS-CoV-2 / Diagnostic Accuracy of Tests Based on Loop-mediated Isothermal Amplification reverse transcriptase (RT-LAMP) for SARS-CoV-2. Perú. Ministerio de Saud. Instituto Nacional de Salud. Lima; Instituto Nacional de Salud. Mayo 2020. Disponible en: http://docs.bvsalud.org/biblioref/2020/07/1116113/rr08_pruebas-rapidas-sars-cov-2-lamp.pdf
- Escalante-Maldonado, Oscar; Vidal-Anzardo, Margot; Donaires, Fernand; Solis-Sanchez, Gilmer; Gallesi, Italo; Pampa-Espinoza, Luis; Huaringa, Maribel; Serrano, Nancy Rojas; García, Coralith; Angles-Yanqui, Eddie; Gavilán, Ronnie Gustavo; Durães-Carvalho, Ricardo; Cabezas, Cesar; Simas, Paulo Vitor Marques. Reverse Transcription-Loop-Mediated Isothermal Amplification (RT-LAMP) is an effective alternative for SARS-CoV-2 molecular detection in middle-income countries. Instituto Nacional de Salud; Octubre 2020. Disponible en: https://doi.org/10.1101/2020.10.14.20212977
- Precisión diagnóstica de pruebas basadas en amplificación isotérmica mediada en lazo de transcriptasa inversa (RTLAMP) para SARS-CoV-2. SERIE REVISIONES RÁPIDAS N° 08-2020. Lima, mayo de 2020. INSTITUTO NACIONAL DE SALUD. http://docs.bvsalud.org/biblioref/2020/07/1116113/rr08_pruebas-rapidas-sars-cov-2-lamp.pdf
- Paz L. Guía para el manejo del COVID-19. :175.
- Paz L. GUÍA DE DIAGNÓSTICO Y TRATAMIENTO DE COVID-19 EN UNIDADES DE TERAPIA INTENSIVA. :115.
- Escalera-Antezana JP, Lizon-Ferrufino NF, Maldonado-Alanoca A, Alarcón-De-la-Vega G, Alvarado-Arnez LE, Balderrama-Saavedra MA, et al. Clinical features of the first cases and a cluster of Coronavirus Disease 2019 (COVID-19) in Bolivia imported from Italy and Spain. Travel Medicine and Infectious Disease. 1 de mayo de 2020;35:101653.
- Escalera-Antezana JP, Cerruto-Zelaya PE, Apaza-Huasco M, Miranda-Rojas SH, Flores-Cárdenas CA, Rivera-Zabala L, et al. Healthcare workers' and students' knowledge regarding the transmission, epidemiology and symptoms of COVID-19 in 41 cities of Bolivia and Colombia. Travel Medicine and Infectious Disease. 1 de septiembre de 2020;37:101702.
- Testing and Screening for COVID-19 | Ministry of Health [Internet]. [citado 22 de julio de 2021]. Disponible en: https://health.gov.tt/covid-19-faqs-and-mythbusters/testing-and-screening-for-covid-19
- CARPHA> Manual de usuario de laboratorio - Servicios de pruebas de laboratorio [Internet]. [citado 22 de julio de 2021]. Disponible en: https://carpha.org/What-We-Do/Laboratory/Laboratory-Testing-Services
- CARPHA. Algorithm for Contact Tracing for Suspected coronavirus disease (COVID-19) cases on conveyances to CARPHA Member States [Internet]. 2020. Disponible en: https://carpha.org/Portals/0/Documents/Technical%20Guidance/Algorithm%20for%20Contact%20Tracing%20for%20Suspected%20coronavirus%20disease%20(COVID-19)%20cases%20on%20conveyances.pdf
- Organización Panamericana de la Salud. Respuesta de la OPS/OMS a la COVID-19 en Paraguay [Internet]. [citado 23 de julio de 2021]. Disponible en: https://iris.paho.org/bitstream/handle/10665.2/53341/OPSPARCOVID-19210002-spa.pdf?sequence=5&isAllowed=y
- Bejarano DP, López E, Duarte J, Grijalba R, González A, Cuenca E, et al. http://scielo.iics.una.py/scielo.php?script=sci_abstract&pid=S2307-33492021000100008&lng=en&nrm=iso&tlng=es. Revista de salud publica del Paraguay. junio de 2021;11(1):8-19.
Recibido: 3 abril 2021
Aceptado: 10 Agosto 2021
Pool Marcos-Carbajal1,2,, Christian Allca-Muñoz14,, Macarena Ganoza-Farro1,4,, André Valdez-Olivera1,4,, Allison Gomez-Martel1,4, , Maribel Huaringa-Nuñez5,, Alberto Salazar-Granara1,3, ,
1. Universidad San Martin de Porres, Facultad de Medicina Humana, Centro de Investigación de Medicina Tradicional y Farmacología, Chiclayo, Perú
2. Universidad Peruana Unión, Escuela Profesional de Medicina Humana, Laboratorio de Investigación en Biología Molecular, Lima, Perú
3. Sociedad Peruana de Farmacología y Terapéutica Experimental – SOPFARTEX, Lima, Perú
4. Sociedad Científica de Estudiantes de Medicina de la Universidad de San Martín de Porres, Lima, Perú
5. Instituto Nacional de Salud, Centro Nacional de Salud Pública, Laboratorio de Virus Respiratorios, Lima, Perú
ORCID:
Pool Marcos Carbajal: https://orcid.org/0000-0002-7741-0337
Alberto Salazar Granara: https://orcid.org/0000-0003-1996-3176
Maribel Huaringa Nuñez: https://orcid.org/0000-0001-9247-4040
Christian Allca Muñoz: https://orcid.org/0000-0001-7381-5823
Macarena Ganoza Farro: https://orcid.org/0000-0003-0296-3989
André Valdez Olivera: https://orcid.org/0000-0002-1002-1425
Allison Gomez Martel: https://orcid.org/0000-0001-9672-8642
Autor de correspondencia
Nombre: Pool Marcos Carbajal
Correo: [email protected], [email protected]
Dirección: Alameda del corregidor 1531, La molina 15024, Teléfono: (01) 3652300