CS 2018.01.01.11
Files > Conference Series > 2018 > CIBB 2018
Bionatura Conference Series Vol 1. No 1. 2018
Publicación del IV CONGRESO INTERNACIONAL DE BIOTECNOLOGÍA Y BIODIVERSIDAD IV CIBB y XV FORO INTERNACIONAL BANANERO
INVESTIGATION / RESEARCH
Identificación molecular del género Passiflora (Passifloraceae), en la región norte del Ecuador mediante el método DNA Barcoding
Molecular identification of the genus Passiflora (Passifloraceae), in the northern region of Ecuador using the DNA Barcoding method
Marco Cerna1 , Jorge Miño2
Available in : http://dx.doi.org/10.21931/RB/CS/2018.01.01.11
RESUMEN
El género Passiflora o también llamado flor de la pasión, en el Ecuador se tiene registros de 95 especies, en esta investigación se colectaron al azar muestras de especies de este grupo ubicados en la región norte del Ecuador en las provincias de: Pichincha, Santo Domingo, Napo, Tungurahua, Cotopaxi; en pisos altitudinales que van desde los 400 hasta los 2800 m.s.n.m, las muestras fueron identificadas a nivel de especie mediante comparación con vouchers del Herbario Nacional del Ecuador QCNE, para la identificación molecular se extrajo el ADN mediante el método de Doyle y Doyle, seguidamente se amplificó un segmento del gen matK mediante la técnica PCR convencional, los productos PCR fueron secuenciados mediante la técnica Sanger, las secuencias fueron analizadas con las herramientas Blast y Boldsystem para identificar a que especie corresponden, siendo Blast la mejor herramienta para identificación. A continuación se realizó un análisis filogenético utilizando los segmentos del gen matK y la información del segmento rcbL que está disponible en Genbank, se elaboraron 3 árboles uno por cada segmento y uno concatendando los árboles construidos por el segmento matk y el concatenado muestran similitud en la agrupación de especies en concordancia con la morfología.
Palabras Clave: Passiflora, matK, rbcL, ADN Bardcoding
ABSTRACT
The name of the genus Passiflora means flower of passion, in Ecuador exist 95 species of this genus, in this research, we took samples at random in these species of this group present in the northern region of Ecuador which were: Pichincha, Santo Domingo, Napo, Tungurahua, Cotopaxi; in altitudinal floors ranging from 400 to 2800 meters above sea level, the samples we identified at the species level by comparison with vouchers of the National Herbarium of Ecuador QCNE, in the other hand about the molecular identification, the DNA was extracted by the Doyle y Doyle method, then we amplified a segment of the matK gene using the conventional PCR technique, PCR products were sequenced using the Sanger technique, the sequences were analyzed with the Blast and Boldsystem tools to identify which species they correspond to, Blast being the best tool for identification. Next, a phylogenetic analysis was performed using the segments matK gene and rcbL segment information that is available in Genbank, 3 trees were constructed one for each segment and one concatenating the trees constructed by the matk segment and the concatenated one show similarity in accordance with the morphology, while the segment rbcL presented a differences.
Key words: Passiflora, matK, rbcL, ADN Bardcoding
INTRODUCCIÓN
El género Passiflora es el más importante de la familia Passifloraceae, debido a su representatividad y al número de especies que agrupa, estas son nativas del Norte, Centro y Sur América, se distribuyen en zonas tropicales que llegan hasta los 3000 m.s.n.m. 1
Existen 534 nombres de especies aceptadas, con un grado de confiabilidad medio del 81,1 % y 18,9 % con bajo grado de confiabilidad, esto revela la necesidad de revisar el estatus de las especies con un sistema más eficiente como lo es el método DNA Barcoding, el cual utiliza segmentos estandarizados de ADN que tienen una variabilidad suficiente para discriminar a los individuos a nivel de especie. 2
Las especies de este género sufren múltiples hibridaciones debido a que estas se entrecruzan fácilmente y forman híbridos que no se han podido identificar siendo fundamental un análisis filogenético usando datos moleculares y con ello ver las relaciones de parentesco que estas presentan. 3
La identificación a nivel molecular resulta ser indispensable al momento de identificar especies pudiendo establecer nuevos datos taxonómicos; en la actualidad la técnica de DNA Barcoding se utiliza para determinar especies mediante el uso de segmentos de ADN cloroplástico que son matK y rbcL 4
El número de especies de este género se ha incrementado en las últimas décadas; es por eso que en este trabajo se ha planteado como objetivo principal la caracterización a nivel molecular de las especies del género Passiflora, presentes en la zona norte del Ecuador, mediante el uso del segmento matK, para justificar la publicación de nuevas especies o reagrupar aquellas que tienen diferencias morfológicas por su carácter híbrido. 5
MATERIALES Y MÉTODOS
Colección de especies
Antes de realizar la salida al campo, se hizo una visita al Herbario Nacional del Ecuador QCNE ubicado en la ciudad de Quito, con el objetivo de ubicar las especies del género Passiflora colectadas en el Ecuador, conocer su morfología y puntos de colección para determinar su distribución.
Una vez obtenido los datos previamente descritos en el herbario QCNE, se realizó la salida de campo a los puntos marcados con el fin de obtener muestras de cada especie, los datos de colección se registraron en el libro de campo de Marco Cerna a partir del código 3366; a cada muestra se le asignó un número de colección, se registró la siguiente información: número de colección, fecha, datos geopolíticos, Nombre científico, nombre común, ubicación geográfica latitud, longitud, entre otros. Las muestras para obtención de ADN fueron hojas jóvenes, sanas y libres manchas y cicatrices, las muestras colectadas se depositan en fundas de papel kraft, con el respectivo número de colección. 6
Extracción de DNA
Para la extracción de ADN total se obtuvo a partir de muestras frescas, hojas jóvenes de las diferentes especies de Passifloras, colectadas en la zona Norte del Ecuador, para ello se realizó el protocolo de extracción simple de ADN foliar de Doyle y Doyle (1987), se colocaron 40 g de tejido foliar fresco en tubos eppendorf de 2 mL, previamente etiquetados con los números de referencia que se obtuvieron durante la recolección; a estas muestras se las pulverizó con la ayuda del nitrógeno líquido. A continuación se agregó 600 µL de Buffer de Lisis (100 mM TrisCl (pH 8), 50 mM EDTA (pH 8) y 500 mM NaCl) y se agitó con la ayuda de un vórtex para homogenizar la muestra, seguidamente se añadió 94 µL de SDS (Dodecilsulfato sódico) al 10 % y se mezcló por inversión 6 veces, posteriormente se procede a colocar los tubos eppendorf en un baño María durante 10 min a 65 ºC, inmediatamente se agregó 118.5 µL de acetato de potasio (K-acetato 5 M), se mezcló por inversión 6 veces, una vez homogenizada la muestra se colocó los tubos en hielo durante 5 minutos. Posteriormente se centrifugó a 13000 rpm por 5 min, en seguida se procede a transferir el sobrenadante a un nuevo tubo eppendorf de 2 mL previamente etiquetados con los números de referencia, luego se adicionó 600 µL de fenol: cloroformo en proporción (1:1), se centrifugó a 13000 rpm por 5 min, después de este proceso se formaron dos fases, la fase superior se transfirió a un nuevo tubo eppendorf de 1.5 mL y finalmente se agregó 360 µL de isopropanol frío, se las mezcló suavemente por inversión 6 veces y se almacenó a-20º C por toda la noche.
Al siguiente día se procedió a centrifugar por 3 minutos a 13000 rpm, después de lo cual se formó un pellet, se eliminó el sobrenadante y luego se lavó con etanol frío al 70 %, se añadió 400 µL de etanol frio, se centrifugó por 3 min a 13000 rpm y se descartó el sobrenadante, se realizó este proceso por tres veces. Para eliminar el solvente mezclado con el pellet se ubicó las muestras en un termo bloque a 37 °C durante 30 min. Finalmente, se re suspendió el pellet en 30 µL de TE 1X (10 mM TrisCl (pH 8) y 1 mM EDTA) que permite conservar el ADN obtenido. 7
Visualización del ADN extraído por medio de electroforesis horizontal
Para determinar la presencia de ADN, mediante la técnica de Electroforesis horizontal se preparó el gel de agarosa al 1 % (50 mL solución TBE 1x y 0.5 g de agarosa ultra pura de Invitrogen, calentada en microondas durante 1 min); cuando la temperatura de la agarosa alcanzó 38 °C, se colocó 5 µL de Sybr Safe de Invitrogen; antes de que se solidifique el gel es necesario ponerlo en el molde rectangular con el peine con el fin de marcarse los posillos donde se colocaran las muestras. Inmediatamente se procedió a agregar la solución TBE 1x hasta que cubra totalmente el gel, cuando la cámara estuvo lista se procedió a cargar las muestras, es decir se tomó 4 µL de ADN cloroplástico y 4 µL de tampón de carga “BLU” compuesto por Xileno Cianol (25 mg) y Sucrosa (4 g), en el caso de producto PCR se tomó 4 µL de producto PCR y 4 µL de tampón de carga “BLU”, antes de programar la cámara se preparó la muestra de la siguiente manera: se toman 3.5 µL de Ladder y 4 µL de tampón de carga “BLU” con la finalidad de observar las pares de bases, una vez cargadas las muestras, se procedió a setear la cámara de electroforesis horizontal Labnet a las siguientes condiciones a 90 Voltios por 40 min, visualizándose el resultado en el fotodocumentador Bio-imaging systems. 8
Amplificación y Secuenciación
Una vez extraído el ADN, se amplificó un segmento del gen Maturasa K matK presentando un tamaño de 730 pb; para lo cual se combinaron los siguientes elementos para un volumen total de 25 µL: 12.5 µL 2 x Phire Plant Direct PCR Master Mix, 1.25 µL Disolution Buffer, 0.5 µL primer forward matK_1 RKIM (ACCCAGTCCATCTGGAAATCTTGGTTC), 0.5 µL primer reverse matK (CGTACAGTACTTTTGTGTTTACGAGAG), 1 µl de ADN, 9.25 µL de agua libre de nucleasas; este protocolo se desarrolló usando un bloque frio LightCycler® Centrifuge Adapters - Roche en la cámara de flujo laminar Espectrocom con materiales previamente esterilizados.
Una vez preparada la master mix se usó el termociclador Labnet Multigene con el siguiente programa: inicio de la desnaturalización 95 ºC por un período de 5 min, a continuación 35 ciclos de: 95 ºC por 40 seg, 52.8 ºC para el temperatura de fusión por 30 seg, 72 °C por 1 min para la extensión inicial; al terminar los ciclos se produce la extensión final a 72 °C por 8 min, finalmente la etapa de mantenimiento a 4 °C. 9
La secuenciación de los productos PCR en esta investigación, se realizó por el método de SANGER, en la cual se identifica a los nucleótidos que forman parte de los segmentos amplificados, las muestras fueron enviadas en un volumen de 20 µL cuya concentración de masa fue de 95 ng/µL.
Consulta de base de datos en Boldsystems
Para completar la información filogenética del género se descargó secuencias del segmento matK de especies del genero Passiflora que no están presentes en la zona de colección, estas fueron P. riparia, P. capsularis, P. sexflora, P. adenopoda, P. biflora, P. micropetala, las cuales se encuentran presentes en el Ecuador; adicionalmente se obtuvieron secuencias del segmento rbcL se complementaron con las especies colectadas y amplificadas con el segmento matK y las siguientes especies: P. auriculata, P. foetida, P. micropetala, P. punctata, P.trifasciata, P.vespertilio.
Construcción del árbol filogenético
Para ello se utilizó el software MEGA 6, el primer paso fue alinear todas las secuencias obtenidas mediante el algoritmo Muscle, que identificó secuencias homólogas y permitió establecer el grado de similitud entre muestras analizadas. 10
Se elaboraron tres árboles filogenéticos, el primero usando un segmento del gen matK, el segundo árbol filogenético usando un segmento de gen rbcL y el tercero concatenado los dos segmentos matK y rbcL; usando el algoritmo NeighborJoiningTree, y el modelo de estimación de distancias de secuencias Jukes-Cantor y bootstrap de 1000 réplicas.
RESULTADOS Y DISCUSIÓN
En este trabajo se colectaron 12 especies del género Passiflora, en la región norte del Ecuador, en cinco provincias Pichincha, Cotopaxi, Tungurahua, Santo Domingo de los Tsáchilas y Napo los datos de colección se registraron en el libro de campo de Marco Cerna como se observa en la tabla 1.
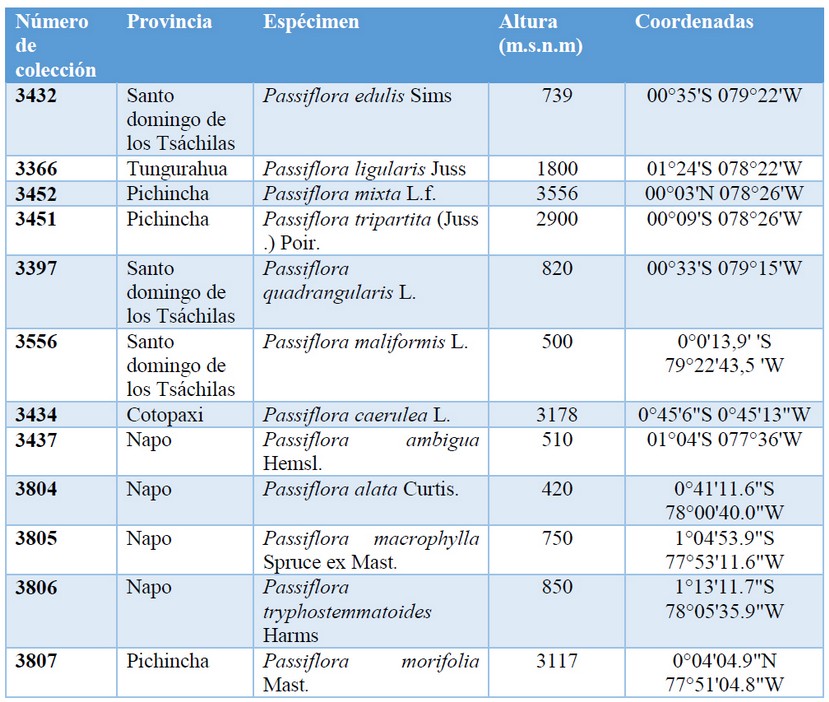
Tabla 1 Listado de las especies colectadas del género Passiflora para este estudio
Identificación molecular
Extracción de ADN
Se obtuvo ADN de las 12 muestras colectadas mediante el método Doyle y Doyle modificado, las cadenas obtenidas tienen una longitud de banda mayor a 5000 pb el tamaño fue determinado mediante comparación con el marcador molecular “Lader O´Gener ruler” con un rango de 0.1 hasta 5 kb, como se muestra en la figura 13 7
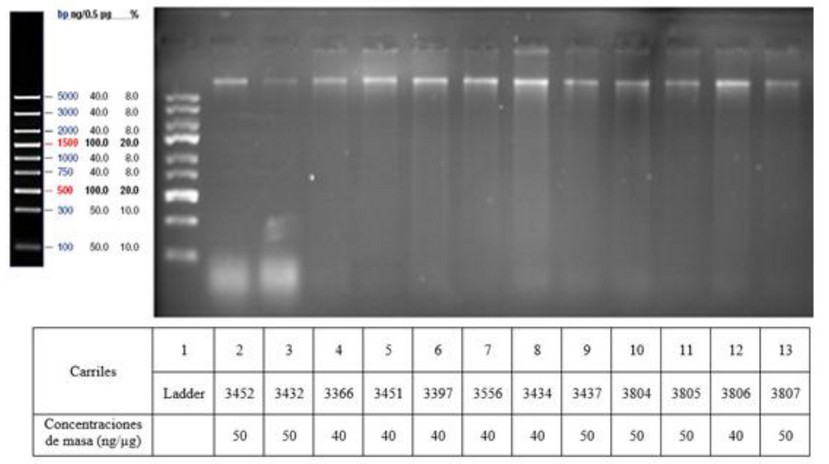
Figura 1 Gel de electroforesis de ADN
Amplificación del segmento matK de las especies colectadas mediante la técnica PCR convencional.
La longitud de las moléculas de ADN presentan un promedio de 765 pb, como se muestra en la figura 14, en comparación con el trabajo de Mazo (2011), referente a estudios de Exsicados de Orquídeas, obtuvo segmentos con 730 pb para el gen matK. 9
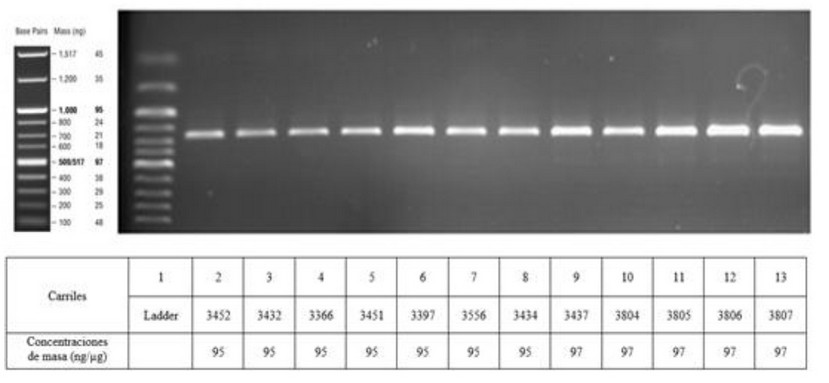
Figura 2 Gel de electroforesis de productos PCR
Identificación de secuencias por medio del software Blast y Boldsystems
Se procedió a identificar las secuencias amplificadas del segmento matK, estas fueron analizadas con el software Blast, como resultado se obtuvo la identidad de las especies: Passiflora edulis, P. quadrangularis, P. actinia, P. caerulea, P. maliformis, P. quadrangularis, P.aurantioides, P. cincinnata y Passiflora spp. con una identidad por encima del 98 %, en contraste con, P. adenopoda y P. gardneri presento una identidad del 94 % siendo muy bajo pero estas especies se encuentran en el mismo género.
Las secuencias también fueron analizadas con el software Boldsystem, el cual no dio resultados iguales o mayores al 98 % de identidad, lo que nos indica que no tiene registros de estas especies en su banco de datos.
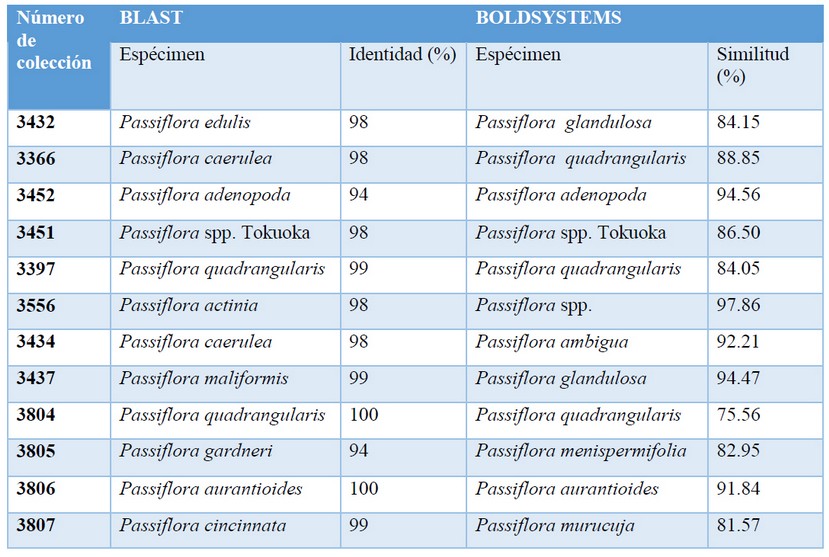
Tabla 2 Resultados de identidad de especies colectadas
Filogenia en base al marcador matK
El árbol filogenético se construyó a partir de 18 secuencias de las cuales 12 fueron especies colectadas y 6 se obtuvieron de la base de datos Gene Bank; como se observa en la figura 15, el árbol muestra cinco clados para el género Passiflora, incluyendo una especie de referencia perteneciente a un grupo externo.
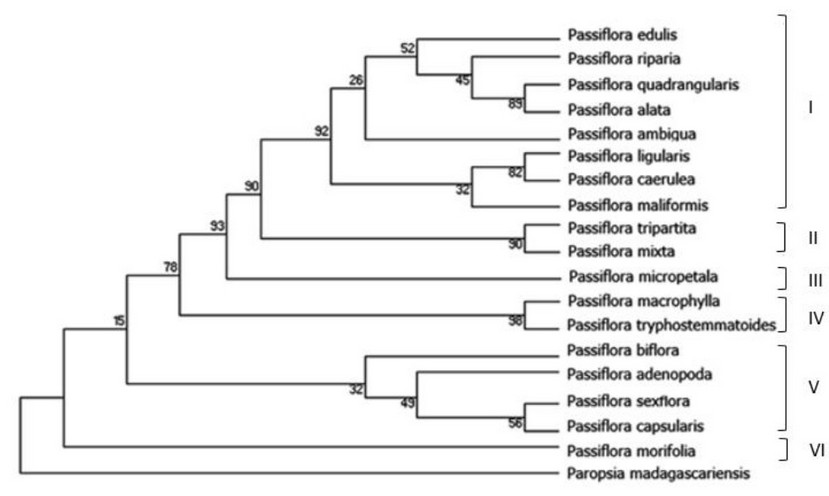
Figura 3 Árbol filogenético mediante el test Neighbor-Joining en la segemento matK
Passiflora biflora y P. adenopoda forma un grupo vecino al clado 1, mientras que en este estudio se los ubica en el grupo IV 12 . Además la especie P. Ambigua la ubican en el cuarto grupo, en este estudio se ubica en el grupo 1. 13
En base a este árbol se determina que el grupo 1 es el más evolucionado mientras que el grupo 5 es el más antiguo o basal, representado por la especie P. morifolia presente en la provincia de Pichincha, esta puede ser la zona de dispersión del género.
Árbol Filogenético por medio del marcador rbcL
El árbol filogenético se construyó con 19 secuencias del marcador rbcL de especies del género Passiflora presentes en Ecuador, estas secuencias fueron descargadas de la Biblioteca virtual Gene Bank; como se observa en la figura 16, el árbol muestra cinco clados, incluye una especie de referencia perteneciente a un grupo externo.
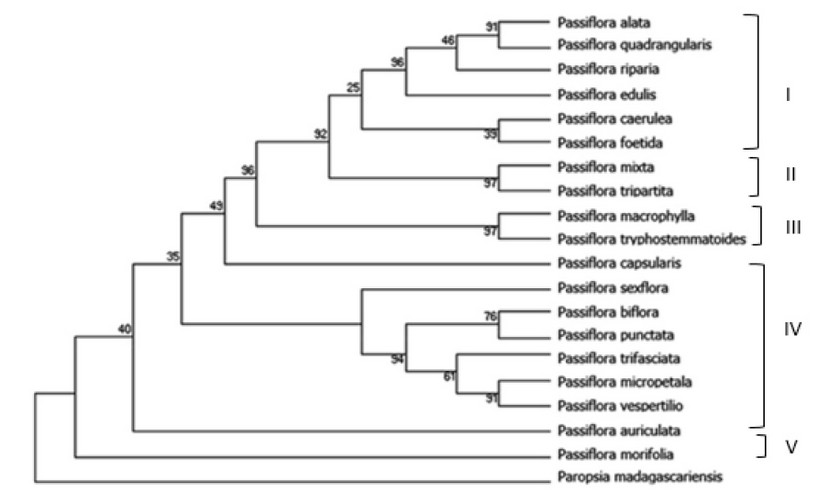
Figura 4 Árbol filogenético mediante el test Neighbor-Joining en la segemento rbcL
P. punctata, P. biflora; P. vespertilio y P. micropetala en el segundo clado en presente trabajo se ubican en el cuarto clado 14. Además P. sexflora y P. capsularis se ubican en cuarto clado, en este trabajo P. capsularis se ubica en un grupo vecino.15
Árbol Filogenético concatenado los marcadores matK y rbcL
El árbol filogenético se construyó a partir de las secuencias del gen matK y rcbL concatenadas, pertenecientes a 14 especies del género Passiflora, como se observa en la figura 17, el árbol muestra cuatro clados, incluye una especie de referencia perteneciente a un grupo externo.
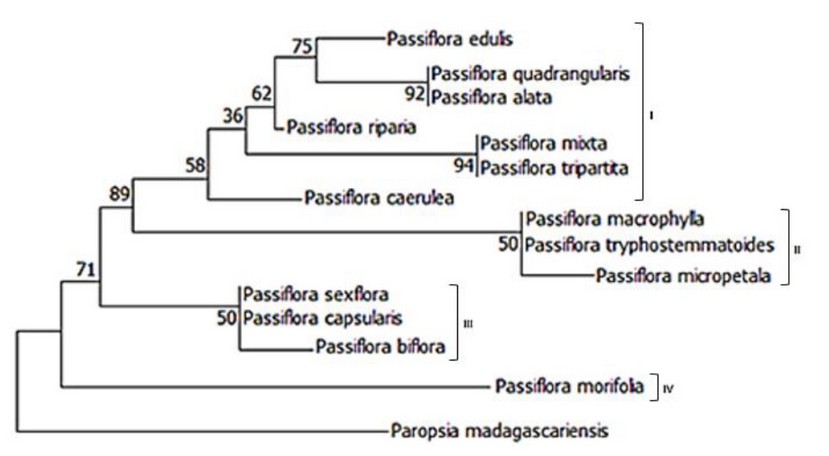
Figura 5 Árbol filogenético mediante el test Neighbor-Joining concatenadno las secuencias matK y rbcL
El árbol concatenado por los segmentos matK y rbcL tiene una conformación similar al árbol construido por el segemento matK, es decir no existe variación en la conformación de grupos, solo presentan ligeros cambios en los soportes de rama, comparando estos resultados con los obtenidos encontramos similitud y coherencia en la distribución de los grupos. 13
Passiflora quadrangularis y P. alata en su morfología son similares, difieren en que las hojas en P. alata presenta en la hoja ocho nervios laterales mientras que P. quadrangularis presenta diez nervios laterales. 16
En segundo lugar, se tuvo el soporte entre P. tripartita y P. mixta la morfología entre las dos especies es bastante similar diferenciándose en los extremos de los pedúnculos y los filamentos del hipantio, es decir a simple vista son idénticos. 17
Por otro lado el soporte entre P. macrophylla y P. tryphostemmatoides son morfológicamente idénticos se diferencian en el tamaño del polen es decir P. macrophylla tiene un tamaño grande de 50 µm mientras que P. tryphostemmatoides tiene un tamaño mediano de 20 µm pero la conformación es la misma oblato esferoidal. 18
Mientras que el soporte entre Passiflora verpertilio y P. micropetala las hace diferentes únicamente la forma de la hoja P. verpertilio presenta la hoja en forma de opuesta en contraste con P. micropetala presenta forma alterna, adicionalmente se tiene que P. biflora y P. punctata presentan pertenecen al mismo subgénero denominado Decaloba, se asemejan en los frutos pequeños y de color purpura. 19
P. morifolia es decir se alejó tempranamente del grupo, su característica que la hace diferente a las demás es el fruto de color azul. 14
CONCLUSIONES
Se colectaron 12 de especies distribuidas en 5 provincias, la mayor abundancia de especies del género Passiflora se encuentra en las regiones cuyos pisos altitudinales van desde entre 400 y 2800 m.s.n.m estas fueran identificadas a nivel de especie, presentan características comunes como son los tallos glabros, zarcillos, semillas planas provistas de una cutícula siendo la característica principal es que todas las semillas son de negro.
Se probó el método de ADN BARCODING para la identificación molecular del género Passiflora siendo un método efectivo y moderno logrando clasificar a las especies de mejor manera mediante el segmento matk presentando muchos polimorfismos permitiendo la discriminación de especies.
El árbol filogenético concatenado elaborado con los segmentos matK y rbcL, estableció clados más precisos, se obtuvo una gran similitud con el árbol realizado por el segmento matK, se denoto que P. morifolia siendo el punto de dispersión del género en la provincia de Pichincha es la especie menos evolucionada mientras que P. edulis, P quadrangularis y P. alata son la especies más moderna del género sus puntos de diversificación son en las zonas tropicales.
REFERENCIAS
1. Cerón C. Bases para el estudio de la flora ecuatoriana. Universitaria, editor. Qutio; 2015. 20-25 p.
2. List TP. The Plant List [Internet]. Version 1.1. 2013. Available from: http://www.theplantlist.org/
3. Hansen A, Gilbert A, Jansen R. Origin and evolutionary relationships in Passrflora. In 1999. p. 73.
4. Krosnick S, Ford A, Freudenstein. Taxonomic Revision of Passiflora subgenus Tetrapathea including the Monotypic Genera Hollrungia and Tetrapathea (Passifloraceae), and a New Species of Passiflora. Systematic Botany; 2009. p. 375–85.
5. Jørgensen P, Muchhala N, MacDougal J. Passiflora unipetala, a new bat-pollinated species of Passiflora supersect. Tacsonia (Passifloracea). Novon; 2012. p. 174–9.
6. Instituto Nacional de Biodiversidad. Protocolo de manejo de colecciones de plantas vasculares proyecto “desarrollando capacidades compartiendo tecnología para la gestión de la biodiversidad en Centroamérica. 2008;5–7.
7. Rivas P, Salazar P. Comparación de la variabilidad genética de tres regiones de ADN cloroplástico y una nuclear en el ishpingo (O. quixos) proveniente de 5 provincias de la Amazonía ecuatoriana [Internet]. Universidad Politécnica Salesiana sede Quito; 2017. Available from: https://www.dspace.ups.edu.ec/bitstream/123456789/4400/1/UPS-ST000985.pdf
8. Lee PY, Costumbrado J, Hsu C-Y, Kim YH. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp [Internet]. 2012;(62):1–5. Available from: http://www.ncbi.nlm.nih.gov/pubmed/22546956
9. Cauz L, Munhoz C, Rodde N, Cauet S, Santos A, Penha H, et al. The Chloroplast Genome of Passiflora edulis (Passifloraceae) Assembled from Long Sequence Reads: Structural Organization and Phylogenomic Studies in Malpighiales. Front Plant Sci [Internet]. 2017;8. Available from: http://journal.frontiersin.org/article/10.3389/fpls.2017.00334/full
10. Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: Molecular evolutionary genetics analysis version 6.0. Mol Biol Evol. 2013;30(12):2725–9.
11. Mazo L. Evaluación Y Comparación De 3 Protocolos De Extracción Y Amplificación Del Adn Contenido En Exsicados De Orquídeas Conservadas En Colecciones De Herbario. Vol. 53. 2011.
12. Tokuoka T. Molecular phylogenetic analysis of Passifloraceae sensu lato (Malpighiales) based on plastid and nuclear DNA sequences. J Plant Res. 2012;
13. Yockteng R, Nadot S. Infrageneric phylogenies: A comparison of chloroplast-expressed glutamine synthetase, cytosol-expressed glutamine synthetase and cpDNA maturase K in Passiflora. Mol Phylogenet Evol. 2004;31(1):397–402.
14. Muschner VC, Lorenz AP, Cervi AC, Bonatto SL, Souza-Chies TT, Salzano FM, et al. A first molecular phylogenetic analysis of Passiflora (Passifloraceae). Am J Bot. 2003;90(8):1235.
15. Bernal N, Ocampo J, Hernández J. Caracterizacion y analisis de la variabilidad genética de la granadilla (Passiflora ligularis juss.) en Colombia empleando marcadores microsatélites. Rev Bras Frutic [Internet]. 2014;36(3):586–97. Available from: http://www.scielo.br/scielo.php?script=sci_arttext&pid=S0100-29452014000300010&lng=es&tlng=es
16. León J. Botánica de los cultivos tropicales. Agroaméric. IICA, editor. San José de Costa Rica; 2000. 139 p.
17. Ocampo J, Coppens d’Eckenbrugge G, Jarvis A, Risterucci A, Feldmann P, Scheldeman X. Study of the genetic diversity of genus Passiflora L.(Passifloraceae) and its distribution in Colombia. 2008.
18. Barrios L. Estudios de la diversidad de passifloraceae en los departamentos de Caldas, Chocó, Nariño, Quindío, Risaralda y Valle del Cauca (Colombia), apoyado en los análisis ecogeográficos, palinológicos y citogenéticos. Universidad Nacional de Colombia; 2005.
19. Esquerre B, Rojas C, Llatas S, Delgado G. El género Passiflora L.(Passifloraceae) en el Departamento de Lambayeque, Perú. [Internet]. Chiclayo; 2014. p. 65. Available from: http://www.biolveg.uma.es/abm/Volumenes/vol39/39_Esquerre-Ibanez_et_al.pdf
Marco Cerna1, Jorge Miño2
1 Dirección de carrera de Biotecnología de los Recursos Naturales sede Quito. Universidad Politécnica Salesiana. Autor de correspondencia. [email protected]
2 Universidad Politécnica Salesiana. [email protected]
