2023.08.04.27
Files > Volume 8 > Vol 8 no 4 2023
Cribado virtual y análisis de complejos enzima-ligando para el factor Xa (FXa) en la cascada de la coagulación humana
Virtual Screening and Analysis of Enzyme-Ligand Complexes for Factor Xa (FXa) in the Human Coagulation Cascade
Virtual Screening and Analysis of Enzyme-Ligand Complexes for Factor Xa (FXa) in the Human Coagulation Cascade
Fabián Santana-Romo 1,2,*, Flavia C. Zacconi 2,3
1 Universidad de las Fuerzas Armadas - ESPE, Departamento de Ciencias Exactas, Sangolquí, Ecuador; [email protected]
2 Pontificia Universidad Católica de Chile, Facultad de Química y de Farmacia, Santiago 7820436, Chile; [email protected] .
3 Pontificia Universidad Católica de Chile, Institute for Biological and Medical Engineering, Schools of Engineering, Medicine and Biological Sciences, Santiago 7820436, Chile; [email protected] .
* Correspondence: [email protected] ; Tel.: (+593 032810206 ext. 4142)
Available from: http://dx.doi.org/10.21931/RB/2023.08.04.27
2 Pontificia Universidad Católica de Chile, Facultad de Química y de Farmacia, Santiago 7820436, Chile; [email protected] .
3 Pontificia Universidad Católica de Chile, Institute for Biological and Medical Engineering, Schools of Engineering, Medicine and Biological Sciences, Santiago 7820436, Chile; [email protected] .
* Correspondence: [email protected] ; Tel.: (+593 032810206 ext. 4142)
Available from: http://dx.doi.org/10.21931/RB/2023.08.04.27
RESUMEN
Esta investigación se centra en el análisis de la información reportada por el Banco de Datos de Proteínas RCSB (RCSB PDB) para la diana molecular del Factor Xa (FXa), una enzima clave en la cascada de la coagulación humana. Este proceso biocatálisis llamado cascada comprende trece factores, proteínas y cofactores activados consecutivamente en reacciones de tipo dominó. El modelo de cascada se propone sobre la base de dos vías de activación: la vía intrínseca iniciada por estímulos procedentes de la superficie dañada del epitelio del vaso sanguíneo y la vía extrínseca iniciada por un traumatismo del tejido del vaso sanguíneo. Estas dos vías convergen en un punto estándar Factor X (FX), cuya finalidad es convertir los monómeros de fibrinógeno en una red de fibrina insoluble, aglutinando el material sanguíneo, los hematíes y las plaquetas para formar un coágulo estable que detenga la hemorragia provocada.
Palabras clave: PDB; FXa; enzima; ligando; informes; cristales; estadística.
Palabras clave: PDB; FXa; enzima; ligando; informes; cristales; estadística.
ABSTRACT
This research focuses on analyzing the information reported by the RCSB Protein Data Bank (RCSB PDB) for the molecular target of Factor Xa (FXa), a key enzyme in the human coagulation cascade. This biocatalytic process called cascade comprises thirteen factors, proteins, and cofactors activated consecutively in domino-type reactions. The cascade model is proposed based on two activation pathways: the intrinsic pathway initiated by stimuli from the damaged surface of the blood vessel epithelium and the extrinsic pathway initiated by trauma to the blood vessel tissue. These two pathways converge at a standard point Factor X (FX), whose purpose is to convert fibrinogen monomers into an insoluble fibrin network, agglutinating blood material, red blood cells, and platelets to form a stable clot that stops the caused bleeding.
Keywords: PDB; FXa; enzyme; ligand; reports; crystals; statistics.
Keywords: PDB; FXa; enzyme; ligand; reports; crystals; statistics.
INTRODUCCIÓN
La hemostasia puede ser definida como el conjunto de procesos biológicos, precisamente integrados, cuya finalidad es mantener la estabilización del coágulo, proteger la integridad del sistema vascular después de una lesión tisular, siendo denominada hemostasia natural estática, y obturando la pérdida del flujo sanguíneo que se produzca en los vasos denominada hemostasia natural correctora. La hemostasia natural tiende a provocar la formación de un coágulo resistente que evite la pérdida de la sangre. Además, la hemostasia efectiva depende de unas complejas interacciones entre: pared vascular, plaquetas, proteínas o factores plasmáticos, incluyendo sus respectivos cofactores.1,2
Cuando se inicia la producción de un coágulo, la formación continua sólo tiene lugar si la sangre no circula, ya que el fluido en movimiento arrastra la trombina y otros agentes procoagulantes liberados durante el proceso de coagulación; alejándolos tan rápidamente que su concentración no logra aumentar efectivamente y promover una mayor coagulación. Por este motivo, la extensión del coágulo casi siempre se interrumpe cuando entra en contacto con la sangre que está fluyendo a una mayor velocidad.3,4
En la década de 1960 se propuso el modelo de cascada de coagulación, este modelo de coagulación introdujo el concepto de que los factores de coagulación existen de una forma "inactiva" denominados zimógenos, o procoagulante, y de una forma "activa". En este último estado, un factor activa al siguiente de forma secuencial con efecto tandem/dominó. El proceso de activación, para la mayoría de los factores, se lleva a cabo por la escisión de una pequeña parte de la forma inactiva, en procesos catalizados por enzimas tal como se muestra en la Tabla 1.

Tabla 1. Descripción de los factores involucrados en el modelo tipo cascada para la coagulación.
Además, se determinó que el factor X (FX) es el punto de convergencia de las vías extrínseca e intrínseca, la primera es activada por estímulos exógenos a la sangre como el factor tisular (FT), mientras que la intrínseca es más bien un sistema consecutivo de activación de proenzimas/zimógenos, postulando que es una proteína determinante como diana terapéutica para el desarrollo de potenciales fármacos que inhiban con efectividad y seguridad el proceso de la coagulación. Es importante mencionar que la denominada cascada de coagulación involucra una serie de cambios bioquímicos y enzimáticos para la formación de trombina y subsecuentemente la formación de un coágulo estable de fibrina.6
El FX es una glicoproteína vitamino K dependiente producida por el hígado. Esta circula en el plasma como dímero con una serina activa en el centro de la cadena pesada de 43000 Daltons y 16000 de la cadena liviana, ambos retenidos por un puente disulfuro. La activación del FX ocurre por la vía extrínseca (tromboplastina) y por la vía intrínseca (IXa, VIIa, Ca2+ y fosfolípidos), obteniendo como resultado el FXa.7
El FXa está situado al inicio de la vía común de coagulación, y dado que la cantidad de serina proteasa se amplifica en cada paso de la cadena con efecto dominó, la hipótesis de la inhibición selectiva de factores de la coagulación, por encima de la trombina, se considera una propuesta antitrombótica de uso prometedor.
El sitio activo del FX está formado por cuatro subsitios denominados S1, S2, S3 y S4. La selectividad del FXa para sus ligandos está dada por el subsitio S1 junto al subsitio S4. El subsitio S1 posee un bolsillo aniónico formado por los residuos Asp189, Ser195 y Tyr228.8
El subsitio S4 reporta tres dominios/sitios de unión: 1) Una caja hidrofóbica formada por los residuos Tyr99, Phe174 y Trp215. 2) Un agujero catiónico formado por los residuos Glu97 y Lys96, y 3) Una caja de agua, situada bajo los residuos Thr98, Ile175 y Thr175.8 Por otro lado, el subsitio S2 es pequeño, poco profundo y definido, y está fusionado con el subsitio S4, mientras que, el subsitio S3 está expuesto al disolvente y situado en la periferia del subsitio S1.
De esta forma se plantea describir de forma actualizada, mediante un cribado virtual automatizado con enfoque al FXa para su estudio, con esto se mejorará su entendimiento a través de un mejor análisis de sus variables complementarias abordando sus complejos cristalinos enzima-ligando. Con la ayuda de los procesos de cribado asistidos por asistencia artificial predictiva se pueden obtener resultados más personalizados enfocados al FXa como objetivo molecular en el modelo de coagulación tipo cascada.
MATERIALES AND METODOS
Mediante la utilización de la plataforma del RCSB Protein Data Bank (RCSB PDB), que permite avances en la ciencia y la educación al proporcionar acceso a herramientas para la exploración, visualización y análisis de estructuras 3D, permite determinar experimentalmente de archivos individuales y librerías. Teniendo también Modelos de Estructura computarizada (CSM), de AlphaFold DB, y ModelArchive. Se realizó una búsqueda específica para el objetivo molecular, a través de un cribado virtual con la identificación “FXa”.
En primera instancia se reportó los complejos estructura-ligando para el FXa de forma integral, generando así tablas individuales para su reporte consolidado. Posteriormente, se tomaron en cuenta los siguientes parámetros:
1) Nombre científico del organismo fuente
2) Taxonomía
3) Método experimental
4) Tipo de entidad polimérica
5) Fecha de lanzamiento
6) Resolución de refinamiento
7) Clasificación enzimática
8) Tipo de simetría
9) Clasificación estructural de proteínas.
RESULTADOS
Una vez ingresado a la plataforma de RCSB PDB, y habiendo realizado previamente una profunda revisión del estado del arte se eligió al FXa como el objetivo molecular, y se procedió a ejecutar el cribado virtual, con las palabras clave FXa en el menú búsqueda avanzada, tomando como referencia que la fecha del reporte es 15 de diciembre de 2022, por lo que es la línea base a considerarse en la presente investigación. Se obtuvieron 115 complejos enzima-ligando reportados como complejos cristalizados, los que en una lista global se descargaron a forma de librería, una vez identificados se procedió a tabular la información, asociando el PDB ID de cada complejo a su literatura reportada, como se aprecia de forma organizada en la Tabla 2.
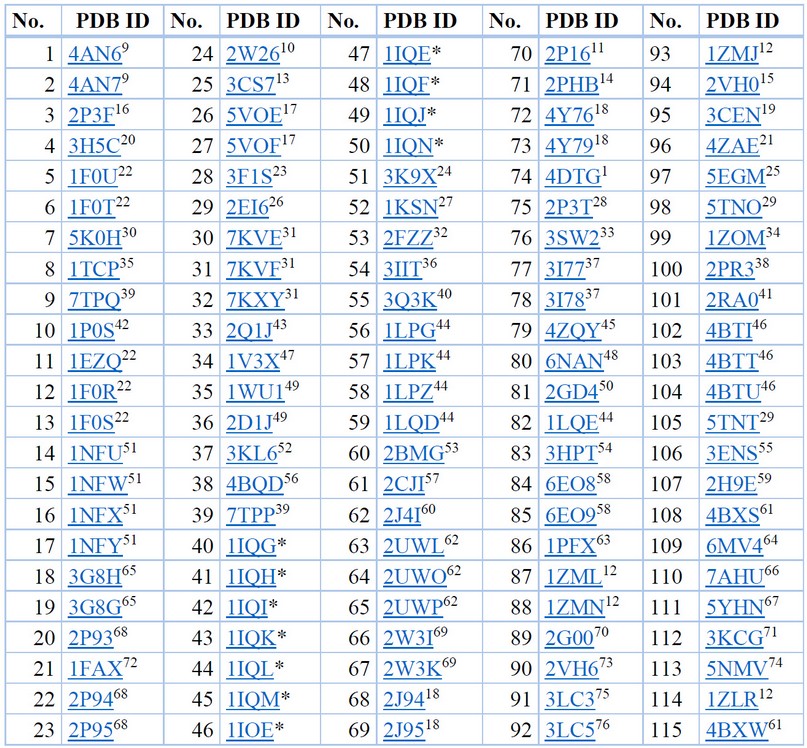
*En proceso de publicación -to be published-. Información recopilada y tabulada.77
Tabla 2. Complejos cristalizados obtenidos a partir del cribado virtual del objetivo molecular del FXa.
Al consolidar la información de todos los cristales reportados para el FXa, se determinó mediante la literatura que las investigaciones tuvieron un alto impacto, ya que generaron cristales obtenidos por el método de difracción de rayos X, siendo así que dentro de una misma publicación se reportan dos o más complejos cristalinos con la enzima del FXa.
Se destaca también que los cristales 4BTI, 4BTT y 4BTU se reportaron como estructuras con fragmentos derivados de aril-piperidina, compuestos diseñados a partir de patrones con su fragmento farmacofórico comprobado a nivel del sitio catalítico (fragmento en color azul). Por otra parte, dentro de la misma investigación se reportaron los cristales 4LOY y 4LXB, con otros blancos farmacológicos diferentes del FXa, se muestra en la Figura 2, los objetivos moleculares en el complejo de cada cristal reportado.46
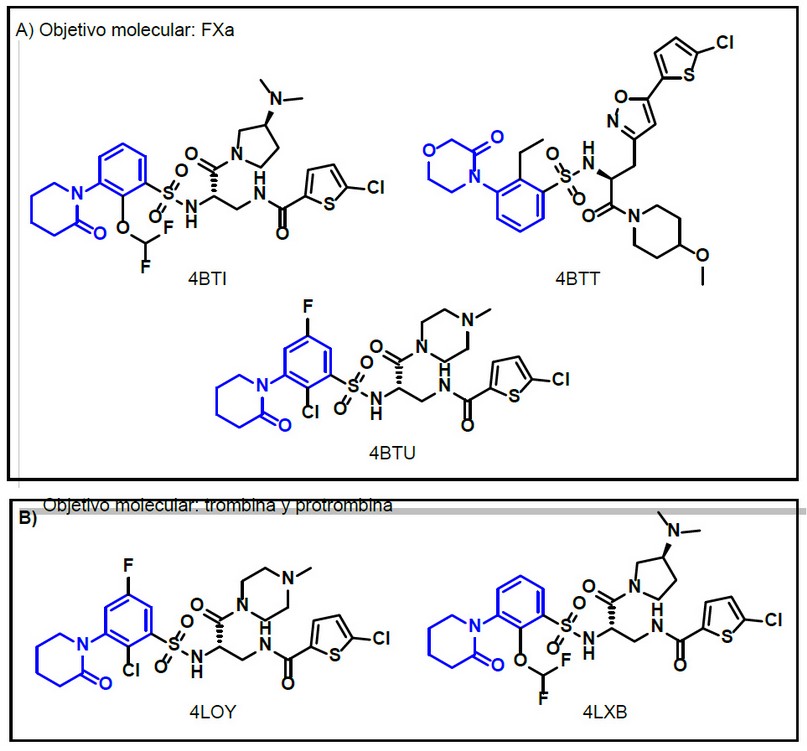
Figura 1. Cristales ensayados con diferentes objetivos moleculares.46
Cristales como 2P16 que corresponde al anticoagulante comercial Apixabán, fármaco: Eliquis® de la casa comercial Pfizer11 ; y 2W26 que corresponde al compuesto anticoagulante Rivaroxabán, fármaco: Xarelto® de la casa comercial Bayer10 . Ambos compuestos se consideran gold standard en el desarrollo de nuevos potenciales anticoagulantes orales, ver Figura 3.
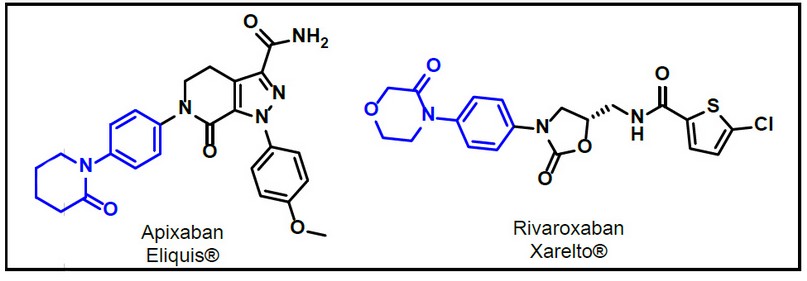
Figura 2. Anticoagulantes gold standard.
Se ha reportado complicaciones posteriores a su suministro en pacientes derivados de cirugías de cadera, por lo que en los últimos 10 años se ha abierto varios expedientes en su uso y seguimiento pre y post cirugía, siendo un hito su prohibición en países europeos como Alemania.78
Adicionalmente, los cristales 7AHU y 7AHV se referencian en la misma literatura, se muestran en la Figura 4, explican que el cristal 7AHU se lo evalúa sobre la enzima del FXa mientras que el cristal 7AHV se contrasta con un anticuerpo biespecífico y mimético del factor VIIa como un estudio complementario a la hemofilia tipo A.66
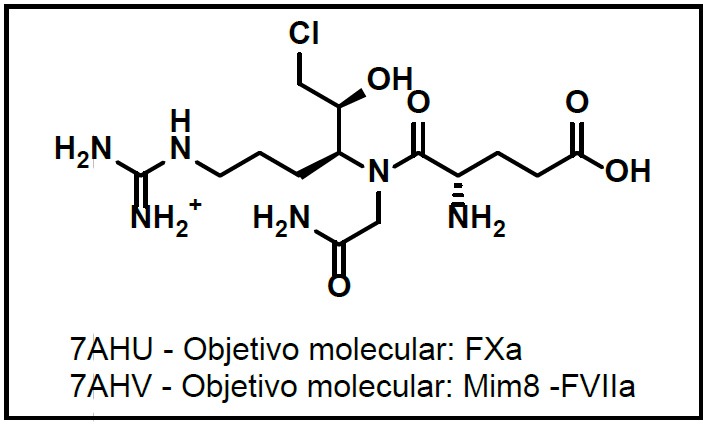
Figura 3. Estructuras ensayadas con multipropósito en variables de coagulación 66
Investigaciones complementarias con enfoque químico teórico y computacional han explicado cómo los gold standard se disponen espacialmente de forma eficiente para lograr la mayor afinidad con el sitio catalítico de la enzima del FXa.79
Estudios de diseño y síntesis basado en procesos cribados automatizados, han permitido crear a través del diseño inteligente nuevos compuestos anticoagulantes con porcentajes de inhibición menor, pero que a su vez proporcionan dosis más seguras y con potencial minimización de efectos adversos.80,81
Nombre científico del organismo fuente
El organismo del que se deriva originalmente la proteína o el nucleótido, se denomina organismo fuente. En algunos casos, estas proteínas y ácidos nucleicos se purifican directamente de las células/tejidos de los organismos de origen. La proteína sigue siendo una proteína humana que se expresa en un huésped bacteriano o en un sistema de expresión. Ver Tabla 3.
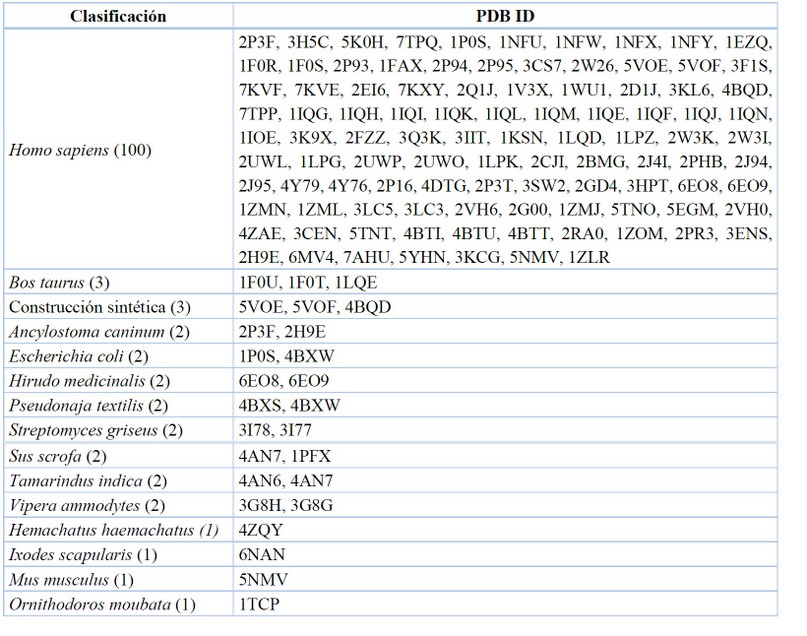
Data recopilada y tabulada.77
Tabla 3. Organismo en la expresión genómica del FXa.
Para efecto de los estudios específicos de los modelos de coagulación, es conveniente trabajar con la expresión génica de Homo sapines, por esa razón se evidencia la cantidad de 100 complejos enzima-ligando reportados en comparación con la expresión en bovinos Bos taurus y construcciones vía síntesis de péptidos.
Taxonomía
El nombre científico jerarquizado de forma taxonómica por el National Center for Biotechnology Information (NCBI) para el organismo de origen asignado por el depositante del RCSB PDB. Para los organismos celulares esto corresponde a un reino (por ejemplo, Archaea, Bacteria, Eukaryota). Para los virus esto corresponde a un clado (por ejemplo, Adnaviria, Bicaudaviridae, Clavaviridae). Para otras entradas y no clasificadas, esto corresponde al primer nivel de cualquier rango taxonómico por debajo del nivel raíz. Ver Tabla 4.
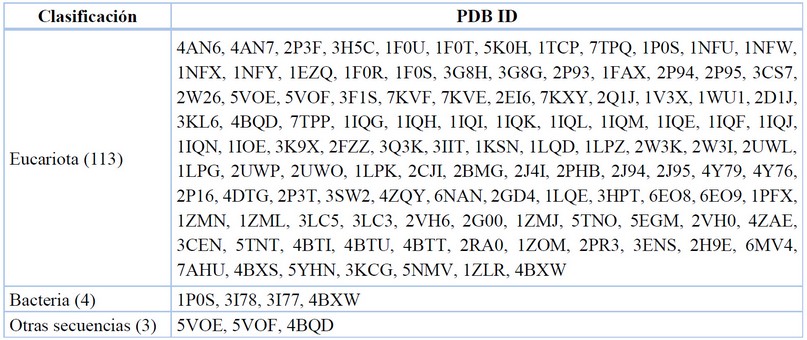
Data recopilada y tabulada.77
Tabla 4. Taxonomía de los organismos reportados del FXa.
Tabla 4. Taxonomía de los organismos reportados del FXa.
Método experimental
La plataforma del RCSB PDB almacena las estructuras en su configuración tridimensional a través de sus coordenadas atómicas (x,y,z). Una estructura resuelta es aquella que ha sido elucidada y reportada mediante una publicación con la caracterización completa de sus heteroátomos.
Existen tres métodos experimentales que permiten determinar la estructura tridimensional de una molécula: 1) a partir de un cristal por difracción de rayos X en disolución, 2) por Resonancia Magnética Nuclear RMN, y 3) a partir de microscopía electrónica por reconstrucción de imágenes. Ver Tabla 5.
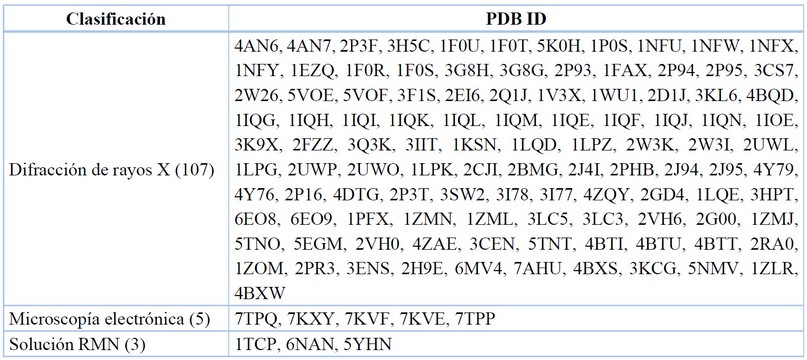
Data recopilada y tabulada.77
Tabla 5. Métodos para elucidar complejos enzima-ligando.
Dentro de la difracción de rayos X, la metodología estándar es el reemplazamiento molecular, que consiste en el empleo de una molécula de estructura conocida para calcular las fases iniciales de una segunda molécula con elementos estructurales comunes con la primera.82
Este es el método comúnmente utilizado para determinar la estructura de macromoléculas como las proteínas, por lo que mediante un estudio preliminar se obtienen y contrastan las coordenadas de los heteroátomos de los aminoácidos constituyentes de las enzimas y se lo corrobora con lo que se obtiene en la determinación experimental de difracción de rayos X; pudiendo establecer una medida de desviación estándar entre lo calculado y reportado. Para el objetivo molecular del FXa, se tienen 107/115 cristales obtenidos mediante la metodología experimental de cristalografía de rayos X.
Tipo de entidad polimérica
En la Tabla 6 se encuentran los cristales reportados en la plataforma del RCSB PDB, que pueden contener múltiples entidades/tipos macromoleculares como:
1) Proteína, cuando una macromolécula muestra claramente su estructura cuaternaria, reflejando así su super enrollamiento molecular,
2) ADN, con su doble hélice de aminoácidos concatenados muestra su estructura terciaria,
3) ARN, con una única hebra/cadena de aminoácidos se usa con fines de expresión de genes,
4) Híbrido, son aquellas posibles combinaciones entre los anteriores,
5) Otro, producto del diseño y síntesis de polímeros entre aminoácidos, monómeros estructurales e inclusive algunos elementos inorgánicos.

Data recopilada y tabulada.77
Tabla 6. Tipo de polímeros orgánicos.
Al realizar el cribado específico para el FXa se pudo obtener los 115 complejos cristalinos reportados en las tablas 2 y 6 respectivamente, todas ellas con relación a los complejos enzima-ligando, diferenciando si la enzima del FXa contiene todas sus cadenas (liviana y pesada) o únicamente la cadena pesada donde se encuentra su sitio catalítico.
Para los cristales 5VOE y 5VOF se utilizan aptámeros de ARN para mejorar la actividad de un anticoagulante alternativo, se demostró que las combinaciones de 11F7t y heparina no fraccionada (HNF) mejoró la actividad anticoagulante.17
Fecha de lanzamiento
El año en que se publicó una entrada de complejo cristalino en la plataforma del RCSB PDB. Sencillamente hace referencia a la literatura reportada en el artículo científico en el que se proponen los diversos ligandos y se los ensaya frente al objetivo molécula en cuestión. La Tabla 8 evidencia el rango de publicación para los complejos enzima-ligando de FXa y que se reportan desde 1995 a 2022.
Para el FXa la primera estructura reportada como complejo enzima-ligando fue el cristal 1TCP, el mismo que tiene dos fechas importantes a diferenciar: 1) fecha de cristalización y reporte de coordenadas el 31 de octubre de 1994, y 2) fecha de publicación del artículo científico (21 de noviembre de 1994) estando disponible para su difusión en 1995.
Los años de mayor apogeo y producción científica en el estudio del FXa se reporta en el intervalo entre 2005 y 2009, teniendo así 44 publicaciones de cristales asociados a este objetivo molecular. Asimismo, los cristales publicados de manera más reciente son 7TPP y 7TPQ, aceptados el 30 de marzo de 2022.39
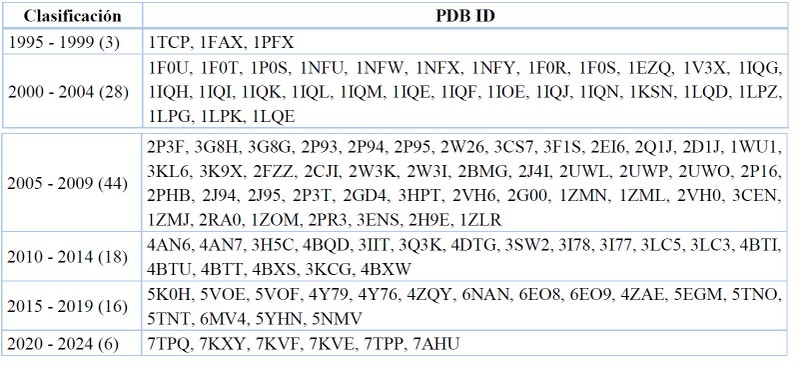
Data recopilada y tabulada.77
Tabla 7. Fecha de lanzamiento para los cristales del FXa.
Resolución de refinamiento (Å)
Los datos mostrados a través de la resolución incluyen estructuras elucidadas por cristalografía de rayos X o microscopía electrónica. Esta estadística se muestra como la desviación cuadrática media raíz (Root Mean Square Deviation) (RMSD) medida en Angstroms (Å), y que indica de forma sencilla que tan fidedigna es una estructura y su reporte de coordenadas con respecto a un patrón teórico. Los rangos reportados para el FXa se muestran en la Tabla 7.
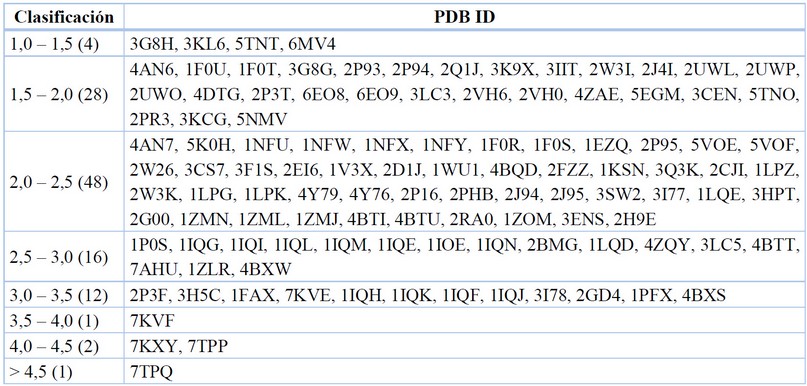
Data recopilada y tabulada.77
Tabla 8. Resolución de proteínas para el FXa.
Tomando en cuenta al objetivo molecular del FXa, la menor resolución reportada fue en 2009 para el cristal 3G8H, con un RMSD de 1,35 Å65 . Mientras que, la mayor resolución reportada tiene un RMSD de 5,30 Å, en el cristal 7TPQ, lo que da a entender una gran diferencia entre las coordenadas teóricas-calculadas y las reportadas en el cristal.39
Para efecto del estudio de los anticoagulantes con mayor relevancia farmacéutica y científica, tenemos al cristal correspondiente a Apixabán, 2P16, con un RMSD de 2,30 Å; y Rivaroxabán, 2W26, con un RMSD de 2,08 Å, lo que indica mayor confiabilidad para su estudio vía acoplamientos moleculares en química teórica computacional.
Clasificación enzimática
La clasificación de enzimas se basa en las recomendaciones del Comité de Nomenclatura de la Unión Internacional de Bioquímica y Biología Molecular (IUBMB). Los números EC (Comisión de Enzimas) se asignan a las estructuras RCSB PDB mediante el mapeo especificado por el autor, UniProtKB GenBank, KEGG. La clasificación para las enzimas del FXa se puede observar en la Tabla 9.
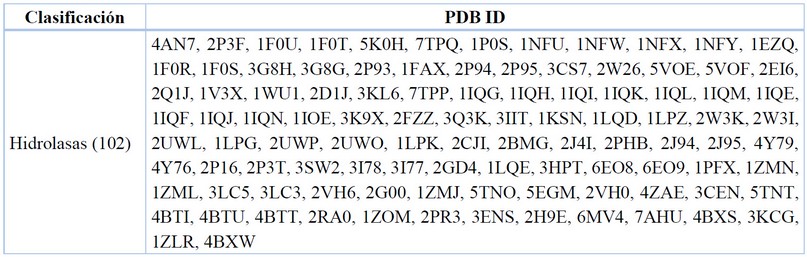
Data recopilada y tabulada.77
Tabla 9. Clasificación enzimática para el FXa.
Una hidrolasa es una enzima capaz de catalizar la hidrólisis de un enlace químico, en el caso del FXa sabemos que en su sitio catalítico ingresará un fragmento de otra proteína, provocando un corte, escindiendo el enlace peptídico en presencia de moléculas de agua como nucleófilos, para generar intermediarios o aductos previos a la separación. Se puede observar que el 100% de los cristales son del tipo hidrolasas, ya que la enzima del FXa, forma parte del grupo de serino proteasas que degradan péptidos y proteínas, y que poseen en fondo de su cavidad catalítica un aminoácido serina esencial para la catálisis enzimática.83
Tipo de simetría
Los ensamblajes oligoméricos se estabilizan mediante interacciones específicas. En algunos casos, los cambios en el entorno o la inclusión de una pequeña molécula o proteína reguladora pueden cambiar las simetrías de ensamblaje. Los ensamblajes de proteínas tienen arreglos simétricos similares y exploran interacciones que conducen a funciones biológicas, cooperatividad y/o regulación. Ver Tabla 10.

Data recopilada y tabulada.77
Tabla 10. Simetría en enzimas para el FXa
La enzima del FXa es una alfa globulina de masa molecular de 58,800 Daltons, compuesta por una cadena liviana de 49 residuos aminoacídicos, además de una cadena pesada con 242 residuos aminoacídicos, las que se unen mediante un puente disulfuro, se destaca que el sitio activo se encuentra como una cavidad periférica en la superficie de esta proteína globular.
De acuerdo con su forma de obtención, purificación y cristalización se las puede encontrar como proteína globular unitaria, dímero, tetrámero. Siendo así las simetrías rotatorias las más comunes, donde se toma muy en cuenta el eje de proyección y giro.84 Teniendo que diferenciar en que subunidad se encuentra el ligando cristalizado, dando el enfoque a esa subunidad para el estudio de las interacciones intermoleculares.85
Clasificación estructural de proteínas
La base de datos Structural Classification of Proteins (SCOP) tiene como objetivo proporcionar una descripción detallada y completa de las relaciones estructurales y evolutivas entre todas las proteínas cuya estructura se conoce. Proporciona un estudio de los pliegues de proteínas, e información sobre la similitud entre proteínas. Como se reporta organizadamente en la Tabla 11.
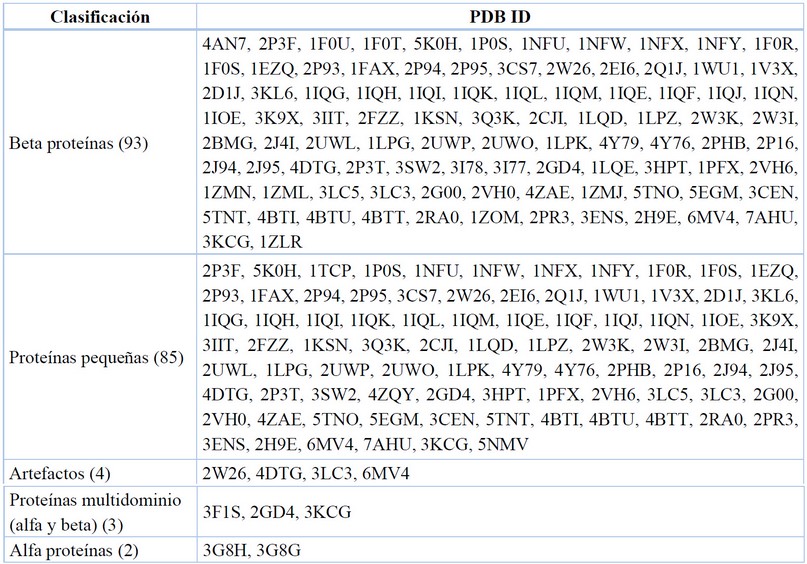
Data recopilada y tabulada.77
Tabla 11. Clasificación SCOP para el FXa.
Las proteínas pequeñas son una clase diversa de proteínas (generalmente <100 aminoácidos de longitud). Su estructura terciaria se mantiene normalmente por puentes disulfuro, ligandos metálicos, y/o interacciones con sus cofactores respectivos.86
La hélice alfa (α-hélice) es común en la estructura secundaria de proteínas donde cada cadena N-H de grupo de enlaces de hidrógeno se enlaza a los grupos C=O del aminoácido situado cuatro residuos antes a lo largo de la secuencia de proteínas.87 La lámina beta (β-hoja) es común en la estructura secundaria de las proteínas. Las láminas beta son hebras conectadas lateralmente, por al menos dos o tres enlaces de hidrógeno de la cadena polimérica, formando una hoja plisada retorcida.88
De esta forma se pudo determinar que la enzima del FXa se reporta mayormente como una beta proteína, por sus interacciones intermoleculares de puente de hidrógeno entre sus residuos aminoacídicos, evidenciándose así las láminas beta. Se reporta también, gran cantidad de proteínas pequeñas, las que no son enzimas completas, pero incluyen en detalle el sitio catalítico para su estudio. Finalmente, se puso en evidencia otros componentes híbridos, los que al ser cristalizados incluyen complementos como cadenas liviana y pesada, además de ligandos alternos y cofactores.
DISCUSIÒN
Los datos presentados fueron interpretados tomando en cuenta el punto de vista químico, adaptando los conocimientos a las aplicaciones biotecnológicas para darle un enfoque médico, utilizando herramientas inter y multidisciplinares, las que, a través de filtros automatizados, como el cribado virtual asistido por computador, nos permitió tener un punto de referencia a diciembre de 2022 con la descripción íntegra del FXa en la plataforma bioinformática del RCSB PDB.
Se establece una línea base, es decir, un antes y un después en las estructuras cristalinas del FXa reportadas en el RSCB PDB, tomando estos datos referenciales para el desarrollo de futuros proyectos de investigación, se enfatiza la obtención de los cristales reportados por difracción de rayos X, contrastando sus resoluciones podemos tener una idea preliminar de calidad y que tan fieles son a sus coordenadas reales.
El fragmento farmacofórico (derivados de aril-piperidina) se obtuvo por estudios preliminares de relación estructura-actividad, los que demostraron que se puede ubicar de forma muy afín en el sitio catalítico, generando las interacciones intermoleculares necesarias para tener actividad frente al FXa, por lo que se puede enunciar que derivados de los mismos (química combinatoria) presentarán bioactividad en esta enzima.
De la totalidad de cristales obtenidos en la plataforma del RCSB PDB con identificación de metadatos referidos al FXa, se encontró que los complejos: 4BTI, 4BTT, 4BTU, 4LOY, y 4LXB se reportaron como estructuras con fragmentos derivados de aril-piperidina, compuestos diseñados a partir de los gold standard comprobando que el diseño racional de fármacos direcciona de forma adecuada a la obtención de nuevos compuestos bioactivos.
Fármacos de referencia como Apixabán (PDB ID: 2P16), con un RMSD de 2,30 Å; y Rivaroxabán (PDB ID: 2W26), con un RMSD de 2,08 Å; son los patrones comerciales a comparar, por su eficiencia como anticoagulantes orales, además, sus complejos cristalinos reportados son los máximos exponentes al momento de realizar estudios de acoplamiento molecular, comparando múltiples propuestas teóricas, con todas las posibilidades en cuanto a disposición espacial como conformeros estructurales. Una vez probados a través de herramientas de química teórica y computacional se procede a su síntesis y evaluación enzimática, a través de pruebas in vitro, de ser muy favorable se procederá a la etapa in vivo, para finalmente, evaluarlo estadísticamente mediante ensayos clínicos.
CONCLUSIONES
Considerando toda la información vertida en este cribado virtual automatizado, se logró generar un análisis a profundidad del objetivo molecular del FXa dentro de la cascada de coagulación humana, describiendo sus variables reportadas, siendo así algunas de ellas cualitativas y las más numerosas cuantitativas. La descripción del FXa permite que a través de este estudio se tenga una línea base actualizada para los reportes de nuevas estructuras cristalinas, su interpretación y utilización en estudios de química teórica y computacional. Así mismo, se plantea la posibilidad de tomar el fragmento farmacofórico descrito para el desarrollo de compuestos bioactivos frente a serino proteasas de características similares al FXa.
Dentro de los datos más representativos se encontró que el cristal 3G8H, con un RMSD de 1,35 Å (menor resolución), en contraste con la mayor resolución reportada tiene un RMSD de 5,30 Å, en el cristal 7TPQ; comparando así la confiabilidad del complejo cristalino en sus coordenadas espaciales reportadas. Estos datos son de vital importancia para la utilización de herramientas químico-computacionales, con fines de investigación y desarrollo en el campo de los compuestos bioactivos.
Author Contributions: Los autores contribuyeron en el presente artículo de investigación bajo el siguiente dfetalle. "Conceptualización, F.S.-R., y F.C.Z.; metodología, F.S.-R., y F.C.Z.; validación, F.S.-R., y F.C.Z.; análisis formal, F.S.-R.; investigación, F.S.-R.; curación de datos, F.S.-R.; redacción—preparación de borrador original, F.S.-R., y F.C.Z.; redacción—revisión y edición F.S.-R., y F.C.Z.; visualización, F.S.-R., y F.C.Z.; supervisión, F.S.-R., y F.C.Z.; administración del proyecto, F.S.-R. Todos los autores han leído y aceptado la versión publicada del manuscrito.
Funding: Esta investigación no recibió financiación externa.
Acknowledgments: F.S.-R., y F.C.Z.; agradecen y dan reconocimiento a ChemAxon por el software MarvinSketch para dibujo químico, visualización, y caracterización de estructuras, subestructuras y reacciones químicas, Marvin 18.27.0 (https://www.chemaxon.com); Instant JChem se utilizó para la gestión, búsqueda y predicción de bases de datos estructuradas, Instant JChem 18.26.0, ChemAxon (http://www.chemaxon.com).
Conflicts of Interest: Los autores declaran no tener conflicto de intereses.
REFERENCES
1. Hilden I, Lauritzen B, Sørensen BB, Clausen JT, Jespersgaard C, Krogh BO, et al. Hemostatic effect of a monoclonal antibody mAb 2021 blocking the interaction between FXa and TFPI in a rabbit hemophilia model. Blood. 2012 Jun 14;119(24):5871–8.
2. Maureane Hoffman. Remodeling the Blood Coagulation Cascade. J Thromb Thrombolysis. 2003;16(1–2):17–20.
3. Panizo Morgado E, Páramo Fernández JA. Fisiología de la hemostasia (2-4) [Internet]. Pediatría Integral 2021; XXV (5): 265.e1 – 265.e11. 2021 [cited 2022 Nov 17]. p. 1–11. Available from: https://www.pediatriaintegral.es/publicacion-2021-07/interpretacion-de-las-pruebas-de-coagulacion/
4. di Minno A, Frigerio B, Spadarella G, Ravani A, Sansaro D, Amato M, et al. Old and new oral anticoagulants: Food, herbal medicines and drug interactions. Blood Rev [Internet]. 2017;1–11. Available from: http://linkinghub.elsevier.com/retrieve/pii/S0268960X16300352
5. Nuñez-Navarro NE, Santana FM, Parra LP, Zacconi FC. Surfing the Blood Coagulation Cascade: Insight into the Vital Factor Xa. Curr Med Chem. 2019;26(17):3175–200.
6. Liesenfeld KH, Lehr T, Dansirikul C, Reilly PA, Connolly SJ, Ezekowitz MD, et al. Population pharmacokinetic analysis of the oral thrombin inhibitor dabigatran etexilate in patients with non-valvular atrial fibrillation from the RE-LY trial. Journal of Thrombosis and Haemostasis. 2011;9(11):2168–75.
7. Pollack C v. Introduction to Direct Oral Anticoagulants and Rationale for Specific Reversal Agents. American Journal of Medicine [Internet]. 2016;129(11):S31–2. Available from: http://dx.doi.org/10.1016/j.amjmed.2016.06.002
8. Abramowitz N, Schechter I, Berger A. On the size of the active site in proteases II. Carboxypeptidase-A. Biochem Biophys Res Commun. 1967 Dec;29(6):862–7.
9. Patil DN, Chaudhary A, Sharma AK, Tomar S, Kumar P. Structural basis for dual inhibitory role of tamarind Kunitz inhibitor (TKI) against factor Xa and trypsin. FEBS Journal. 2012 Dec;279(24):4547–64.
10. Roehrig S, Straub A, Pohlmann J, Lampe T, Pernerstorfer J, Schlemmer KH, et al. Discovery of the Novel Antithrombotic Agent 5-Chloro-N-({(5S)-2-oxo-3- [4-(3-oxomorpholin-4-yl)phenyl]-1,3-oxazolidin-5-yl}methyl)thiophene- 2-carboxamide (BAY 59-7939): An Oral, Direct Factor Xa Inhibitor. J Med Chem. 2005 Sep 1;48(19):5900–8.
11. Pinto DJP, Orwat MJ, Koch S, Rossi KA, Alexander RS, Smallwood A, et al. Discovery of 1-(4-Methoxyphenyl)-7-oxo-6-(4-(2-oxopiperidin-1-yl)phenyl)-4,5,6,7-tetrahydro- 1H-pyrazolo[3,4-c]pyridine-3-carboxamide (Apixaban, BMS-562247), a Highly Potent, Selective, Efficacious, and Orally Bioavailable Inhibitor of Blood Coagulation Factor Xa. J Med Chem. 2007 Nov 1;50(22):5339–56.
12. Lazarova TI, Jin L, Rynkiewicz M, Gorga JC, Bibbins F, Meyers H v., et al. Synthesis and in vitro biological evaluation of aryl boronic acids as potential inhibitors of factor XIa. Bioorg Med Chem Lett. 2006 Oct;16(19):5022–7.
13. Qiao JX, Cheney DL, Alexander RS, Smallwood AM, King SR, He K, et al. Achieving structural diversity using the perpendicular conformation of alpha-substituted phenylcyclopropanes to mimic the bioactive conformation of ortho-substituted biphenyl P4 moieties: Discovery of novel, highly potent inhibitors of Factor Xa. Bioorg Med Chem Lett. 2008 Jul;18(14):4118–23.
14. Kohrt JT, Bigge CF, Bryant JW, Casimiro-Garcia A, Chi L, Cody WL, et al. The Discovery of (2R,4R)-N-(4-chlorophenyl)-N- (2-fluoro-4-(2-oxopyridin-1(2H)-yl)phenyl)-4-methoxypyrrolidine-1,2-dicarboxamide (PD 0348292), an Orally Efficacious Factor Xa Inhibitor. Chem Biol Drug Des. 2007 Aug;70(2):100–12.
15. Young RJ, Borthwick AD, Brown D, Burns-Kurtis CL, Campbell M, Chan C, et al. Structure and property based design of factor Xa inhibitors: Biaryl pyrrolidin-2-ones incorporating basic heterocyclic motifs. Bioorg Med Chem Lett. 2008 Jan;18(1):28–33.
16. Rios-Steiner JL, Murakami MT, Tulinsky A, Arni RK. Active and Exo-site Inhibition of Human Factor Xa: Structure of des-Gla Factor Xa Inhibited by NAP5, a Potent Nematode Anticoagulant Protein from Ancylostoma caninum. J Mol Biol. 2007 Aug;371(3):774–86.
17. Gunaratne R, Kumar S, Frederiksen JW, Stayrook S, Lohrmann JL, Perry K, et al. Combination of aptamer and drug for reversible anticoagulation in cardiopulmonary bypass. Nat Biotechnol. 2018 Aug 4;36(7):606–13.
18. Chan C, Borthwick AD, Brown D, Burns-Kurtis CL, Campbell M, Chaudry L, et al. Factor Xa Inhibitors: S1 Binding Interactions of a Series of N -{(3S)-1-[(1S)-1-Methyl-2-morpholin-4-yl-2-oxoethyl]-2-oxopyrrolidin-3-yl}sulfonamides. J Med Chem. 2007 Apr 1;50(7):1546–57.
19. Corte JR, Fang T, Pinto DJP, Han W, Hu Z, Jiang XJ, et al. Structure–activity relationships of anthranilamide-based factor Xa inhibitors containing piperidinone and pyridinone P4 moieties. Bioorg Med Chem Lett. 2008 May;18(9):2845–9.
20. Huang X, Dementiev A, Olson ST, Gettins PGW. Basis for the Specificity and Activation of the Serpin Protein Z-dependent Proteinase Inhibitor (ZPI) as an Inhibitor of Membrane-associated Factor Xa. Journal of Biological Chemistry. 2010 Jun;285(26):20399–409.
21. Zhang T, Andre P, Bateman TJ, Chen YH, Desai K, Ellsworth K, et al. Development of a novel class of potent and selective FIXa inhibitors. Bioorg Med Chem Lett. 2015 Nov;25(21):4945–9.
22. Maignan S, Guilloteau JP, Pouzieux S, Choi-Sledeski YM, Becker MR, Klein SI, et al. Crystal Structures of Human Factor Xa Complexed with Potent Inhibitors. J Med Chem. 2000 Aug 1;43(17):3226–32.
23. Wei Z, Yan Y, Carrell RW, Zhou A. Crystal structure of protein Z–dependent inhibitor complex shows how protein Z functions as a cofactor in the membrane inhibition of factor X. Blood. 2009 Oct 22;114(17):3662–7.
24. Shi Y, Li C, O’Connor SP, Zhang J, Shi M, Bisaha SN, et al. Aroylguanidine-based factor Xa inhibitors: The discovery of BMS-344577. Bioorg Med Chem Lett. 2009 Dec;19(24):6882–9.
25. Meng D, Andre P, Bateman TJ, Berger R, Chen YH, Desai K, et al. Development of a novel tricyclic class of potent and selective FIXa inhibitors. Bioorg Med Chem Lett. 2015 Nov;25(22):5437–43.
26. Nagata T, Yoshino T, Haginoya N, Yoshikawa K, Isobe Y, Furugohri T, et al. Cycloalkanediamine derivatives as novel blood coagulation factor Xa inhibitors. Bioorg Med Chem Lett. 2007 Aug;17(16):4683–8.
27. Guertin KR, Gardner CJ, Klein SI, Zulli AL, Czekaj M, Gong Y, et al. Optimization of the β-Aminoester class of factor Xa inhibitors. part 2: Identification of FXV673 as a potent and selective inhibitor with excellent In vivo anticoagulant activity. Bioorg Med Chem Lett. 2002 Jun;12(12):1671–4.
28. Ye B, Arnaiz DO, Chou YL, Griedel BD, Karanjawala R, Lee W, et al. Thiophene-Anthranilamides as Highly Potent and Orally Available Factor Xa Inhibitors. J Med Chem. 2007 Jun 1;50(13):2967–80.
29. Sakurada I, Endo T, Hikita K, Hirabayashi T, Hosaka Y, Kato Y, et al. Discovery of novel aminobenzisoxazole derivatives as orally available factor IXa inhibitors. Bioorg Med Chem Lett. 2017 Jun;27(11):2622–8.
30. Steinmetzer T, Sturzebecher J, Schuster O, Sturzebecher U, Schweinitz A, Sturzebecher A. New Substrate Analogue Inhibitors of Factor Xa Containing 4-Amidinobenzylamide as P1 Residue: Part 1. Med Chem (Los Angeles). 2006 Jul 1;2(4):349–61.
31. Ruben EA, Rau MJ, Fitzpatrick JAJ, di Cera E. Cryo-EM structures of human coagulation factors V and Va. Blood. 2021 Jun 3;137(22):3137–44.
32. Pinto DJP, Orwat MJ, Quan ML, Han Q, Galemmo RA, Amparo E, et al. 1-[3-Aminobenzisoxazol-5′-yl]-3-trifluoromethyl-6-[2′-(3-(R)-hydroxy-N-pyrrolidinyl)methyl-[1,1′]-biphen-4-yl]-1,4,5,6-tetrahydropyrazolo-[3,4-c]-pyridin-7-one (BMS-740808) a highly potent, selective, efficacious, and orally bioavailable inhibitor of blood coagulation factor Xa. Bioorg Med Chem Lett. 2006 Aug;16(15):4141–7.
33. Shi Y, O’Connor SP, Sitkoff D, Zhang J, Shi M, Bisaha SN, et al. Arylsulfonamidopiperidone derivatives as a novel class of factor Xa inhibitors. Bioorg Med Chem Lett. 2011 Dec;21(24):7516–21.
34. Lin J, Deng H, Jin L, Pandey P, Quinn J, Cantin S, et al. Design, Synthesis, and Biological Evaluation of Peptidomimetic Inhibitors of Factor XIa as Novel Anticoagulants. J Med Chem. 2006 Dec 1;49(26):7781–91.
35. Lim-Wilby MSL, Vlasuk GP, Brunck TK, Hallenga K, Maeyer M de, Lasters I. NMR structure determination of tick anticoagulant peptide (TAP). Protein Science. 2008 Dec 31;4(2):178–86.
36. Yoshikawa K, Kobayashi S, Nakamoto Y, Haginoya N, Komoriya S, Yoshino T, et al. Design, synthesis, and SAR of cis-1,2-diaminocyclohexane derivatives as potent factor Xa inhibitors. Part II: Exploration of 6–6 fused rings as alternative S1 moieties. Bioorg Med Chem. 2009 Dec 15;17(24):8221–33.
37. Page MJ, di Cera E. Combinatorial Enzyme Design Probes Allostery and Cooperativity in the Trypsin Fold. J Mol Biol. 2010 Jun;399(2):306–19.
38. van Huis CA, Bigge CF, Casimiro-Garcia A, Cody WL, Dudley DA, Filipski KJ, et al. Structure-based Drug Design of Pyrrolidine-1, 2-dicarboxamides as a Novel Series of Orally Bioavailable Factor Xa Inhibitors. Chem Biol Drug Des. 2007 Jun;69(6):444–50.
39. Ruben EA, Summers B, Rau MJ, Fitzpatrick JAJ, di Cera E. Cryo-EM structure of the prothrombin-prothrombinase complex. Blood. 2022 Jun 16;139(24):3463–73.
40. Yoshikawa K, Yoshino T, Yokomizo Y, Uoto K, Naito H, Kawakami K, et al. Design, synthesis and SAR of novel ethylenediamine and phenylenediamine derivatives as factor Xa inhibitors. Bioorg Med Chem Lett. 2011 Apr;21(7):2133–40.
41. Lee YK, Parks DJ, Lu T, Thieu T v., Markotan T, Pan W, et al. 7-Fluoroindazoles as Potent and Selective Inhibitors of Factor Xa. J Med Chem. 2008 Jan 1;51(2):282–97.
42. Wang SX, Hur E, Sousa CA, Brinen L, Slivka EJ, Fletterick RJ. The Extended Interactions and Gla Domain of Blood Coagulation Factor Xa. Biochemistry. 2003 Jul 1;42(26):7959–66.
43. Kohrt JT, Filipski KJ, Cody WL, Bigge CF, La F, Welch K, et al. The discovery of glycine and related amino acid-based factor Xa inhibitors. Bioorg Med Chem. 2006 Jul;14(13):4379–92.
44. Matter H, Defossa E, Heinelt U, Blohm PM, Schneider D, Müller A, et al. Design and Quantitative Structure−Activity Relationship of 3-Amidinobenzyl-1H-indole-2-carboxamides as Potent, Nonchiral, and Selective Inhibitors of Blood Coagulation Factor Xa. J Med Chem. 2002 Jun 1;45(13):2749–69.
45. Barnwal B, Jobichen C, Girish VM, Foo CS, Sivaraman J, Kini RM. Ringhalexin from Hemachatus haemachatus: A novel inhibitor of extrinsic tenase complex. Sci Rep. 2016 Sep 13;6(1):25935.
46. Meneyrol J, Follmann M, Lassalle G, Wehner V, Barre G, Rousseaux T, et al. 5-Chlorothiophene-2-carboxylic Acid [(S)-2-[2-Methyl-3-(2-oxopyrrolidin-1-yl)benzenesulfonylamino]-3-(4-methylpiperazin-1-yl)-3-oxopropyl]amide (SAR107375), a Selective and Potent Orally Active Dual Thrombin and Factor Xa Inhibitor. J Med Chem. 2013 Dec 12;56(23):9441–56.
47. Haginoya N, Kobayashi S, Komoriya S, Yoshino T, Suzuki M, Shimada T, et al. Synthesis and Conformational Analysis of a Non-Amidine Factor Xa Inhibitor That Incorporates 5-Methyl-4,5,6,7-tetrahydrothiazolo[5,4-c]pyridine as S4 Binding Element. J Med Chem. 2004 Oct 1;47(21):5167–82.
48. de Paula VS, Sgourakis NG, Francischetti IMB, Almeida FCL, Monteiro RQ, Valente AP. NMR structure determination of Ixolaris and factor X(a) interaction reveals a noncanonical mechanism of Kunitz inhibition. Blood. 2019 Aug 22;134(8):699–708.
49. Komoriya S, Haginoya N, Kobayashi S, Nagata T, Mochizuki A, Suzuki M, et al. Design, synthesis, and biological activity of non-basic compounds as factor Xa inhibitors: SAR study of S1 and aryl binding sites. Bioorg Med Chem. 2005 Jun;13(12):3927–54.
50. Johnson DJD, Li W, Adams TE, Huntington JA. Antithrombin–S195A factor Xa-heparin structure reveals the allosteric mechanism of antithrombin activation. EMBO J. 2006 May 3;25(9):2029–37.
51. Maignan S, Guilloteau JP, Choi-Sledeski YM, Becker MR, Ewing WR, Pauls HW, et al. Molecular Structures of Human Factor Xa Complexed with Ketopiperazine Inhibitors: Preference for a Neutral Group in the S1 Pocket. J Med Chem. 2003 Feb 1;46(5):685–90.
52. Fujimoto T, Imaeda Y, Konishi N, Hiroe K, Kawamura M, Textor GP, et al. Discovery of a Tetrahydropyrimidin-2(1 H )-one Derivative (TAK-442) as a Potent, Selective, and Orally Active Factor Xa Inhibitor. J Med Chem. 2010 May 13;53(9):3517–31.
53. Matter H, Will DW, Nazaré , Marc, Schreuder H, Laux V, Wehner V. Structural Requirements for Factor Xa Inhibition by 3-Oxybenzamides with Neutral P1 Substituents: Combining X-ray Crystallography, 3D-QSAR, and Tailored Scoring Functions. J Med Chem. 2005 May 1;48(9):3290–312.
54. Shi Y, Zhang J, Shi M, O’Connor SP, Bisaha SN, Li C, et al. Cyanoguanidine-based lactam derivatives as a novel class of orally bioavailable factor Xa inhibitors. Bioorg Med Chem Lett. 2009 Aug;19(15):4034–41.
55. Shi Y, Sitkoff D, Zhang J, Klei HE, Kish K, Liu ECK, et al. Design, Structure−Activity Relationships, X-ray Crystal Structure, and Energetic Contributions of a Critical P1 Pharmacophore: 3-Chloroindole-7-yl-Based Factor Xa Inhibitors. J Med Chem. 2008 Dec 11;51(23):7541–51.
56. Dockal M, Hartmann R, Fries M, Thomassen MCLGD, Heinzmann A, Ehrlich H, et al. Small Peptides Blocking Inhibition of Factor Xa and Tissue Factor-Factor VIIa by Tissue Factor Pathway Inhibitor (TFPI). Journal of Biological Chemistry. 2014 Jan;289(3):1732–41.
57. Watson NS, Brown D, Campbell M, Chan C, Chaudry L, Convery MA, et al. Design and synthesis of orally active pyrrolidin-2-one-based factor Xa inhibitors. Bioorg Med Chem Lett. 2006 Jul;16(14):3784–8.
58. Belviso BD, Caliandro R, de Candia M, Zaetta G, Lopopolo G, Incampo F, et al. How a β-D-Glucoside Side Chain Enhances Binding Affinity to Thrombin of Inhibitors Bearing 2-Chlorothiophene as P1 Moiety: Crystallography, Fragment Deconstruction Study, and Evaluation of Antithrombotic Properties. J Med Chem. 2014 Oct 23;57(20):8563–75.
59. Murakami MT, Rios-Steiner J, Weaver SE, Tulinsky A, Geiger JH, Arni RK. Intermolecular Interactions and Characterization of the Novel Factor Xa Exosite Involved in Macromolecular Recognition and Inhibition: Crystal Structure of Human Gla-domainless Factor Xa Complexed with the Anticoagulant Protein NAPc2 from the Hematophagous Nematode Ancylostoma caninum. J Mol Biol. 2007 Feb;366(2):602–10.
60. Young RJ, Campbell M, Borthwick AD, Brown D, Burns-Kurtis CL, Chan C, et al. Structure- and property-based design of factor Xa inhibitors: Pyrrolidin-2-ones with acyclic alanyl amides as P4 motifs. Bioorg Med Chem Lett. 2006 Dec;16(23):5953–7.
61. Lechtenberg BC, Murray-Rust TA, Johnson DJD, Adams TE, Krishnaswamy S, Camire RM, et al. Crystal structure of the prothrombinase complex from the venom of Pseudonaja textilis. Blood. 2013 Oct 17;122(16):2777–83.
62. Young RJ, Brown D, Burns-Kurtis CL, Chan C, Convery MA, Hubbard JA, et al. Selective and dual action orally active inhibitors of thrombin and factor Xa. Bioorg Med Chem Lett. 2007 May;17(10):2927–30.
63. Brandstetter H, Bauer M, Huber R, Lollar P, Bode W. X-ray structure of clotting factor IXa: active site and module structure related to Xase activity and hemophilia B. Proceedings of the National Academy of Sciences. 1995 Oct 10;92(21):9796–800.
64. Vadivel K, Schreuder HA, Liesum A, Schmidt AE, Goldsmith G, Bajaj SP. Sodium‐site in serine protease domain of human coagulation factor IX a: evidence from the crystal structure and molecular dynamics simulations study. Journal of Thrombosis and Haemostasis. 2019 Apr 6;17(4):574–84.
65. Saul FA, Prijatelj-Žnidaršič P, Vulliez-le Normand B, Villette B, Raynal B, Pungerčar J, et al. Comparative structural studies of two natural isoforms of ammodytoxin, phospholipases A2 from Vipera ammodytes ammodytes which differ in neurotoxicity and anticoagulant activity☆. J Struct Biol. 2010 Mar;169(3):360–9.
66. Østergaard H, Lund J, Greisen PJ, Kjellev S, Henriksen A, Lorenzen N, et al. A factor VIIIa–mimetic bispecific antibody, Mim8, ameliorates bleeding upon severe vascular challenge in hemophilia A mice. Blood. 2021 Oct 7;138(14):1258–68.
67. Ramesh K, Lama D, Tan KW, Nguyen VS, Chew FT, Verma CS, et al. Homologous Lympho-Epithelial Kazal-type Inhibitor Domains Delay Blood Coagulation by Inhibiting Factor X and XI with Differential Specificity. Structure. 2018 Sep;26(9):1178-1186.e3.
68. Qiao JX, Chang CH, Cheney DL, Morin PE, Wang GZ, King SR, et al. SAR and X-ray structures of enantiopure 1,2-cis-(1R,2S)-cyclopentyldiamine and cyclohexyldiamine derivatives as inhibitors of coagulation Factor Xa. Bioorg Med Chem Lett. 2007 Aug;17(16):4419–27.
69. van Huis CA, Casimiro-Garcia A, Bigge CF, Cody WL, Dudley DA, Filipski KJ, et al. Exploration of 4,4-disubstituted pyrrolidine-1,2-dicarboxamides as potent, orally active Factor Xa inhibitors with extended duration of action. Bioorg Med Chem. 2009 Mar;17(6):2501–11.
70. Pinto DJP, Galemmo RA, Quan ML, Orwat MJ, Clark C, Li R, et al. Discovery of potent, efficacious, and orally bioavailable inhibitors of blood coagulation factor Xa with neutral P1 moieties. Bioorg Med Chem Lett. 2006 Nov;16(21):5584–9.
71. Johnson DJD, Langdown J, Huntington JA. Molecular basis of factor IXa recognition by heparin-activated antithrombin revealed by a 1.7-Å structure of the ternary complex. Proceedings of the National Academy of Sciences. 2010 Jan 12;107(2):645–50.
72. Brandstetter H, Kühne A, Bode W, Huber R, von der Saal W, Wirthensohn K, et al. X-ray Structure of Active Site-inhibited Clotting Factor Xa. Journal of Biological Chemistry. 1996 Nov;271(47):29988–92.
73. Young RJ, Borthwick AD, Brown D, Burns-Kurtis CL, Campbell M, Chan C, et al. Structure and property based design of factor Xa inhibitors: Pyrrolidin-2-ones with biaryl P4 motifs. Bioorg Med Chem Lett. 2008 Jan;18(1):23–7.
74. Augustsson C, Svensson A, Kjær B, Chao T ‐Y., Wenjuan X, Krogh BO, et al. Factor Xa and VIIa inhibition by tissue factor pathway inhibitor is prevented by a monoclonal antibody to its Kunitz‐1 domain. Journal of Thrombosis and Haemostasis. 2018 May 6;16(5):893–904.
75. Wang S, Beck R, Blench T, Burd A, Buxton S, Malic M, et al. Studies of Benzothiophene Template as Potent Factor IXa (FIXa) Inhibitors in Thrombosis. J Med Chem. 2010 Feb 25;53(4):1465–72.
76. Wang S, Beck R, Burd A, Blench T, Marlin F, Ayele T, et al. Structure Based Drug Design: Development of Potent and Selective Factor IXa (FIXa) Inhibitors. J Med Chem. 2010 Feb 25;53(4):1473–82.
77. RCSB Protein Data Bank. RCSB Protein Data Bank - Query: ‘FXa’ [Internet]. 2022 [cited 2022 Dec 14]. Available from: https://www.rcsb.org/
78. Steiner T, Böhm M, Dichgans M, Diener HC, Ell C, Endres M, et al. Recommendations for the emergency management of complications associated with the new direct oral anticoagulants (DOACs), apixaban, dabigatran and rivaroxaban. Clinical Research in Cardiology. 2013 Jun 14;102(6):399–412.
79. Durán R, Núñez-Navarro N, Zacconi FC, Herrera B. Theoretical study of C-arylations with aryl halides to determine the reaction mechanism, the effect of substituents and heteroatoms. Physical Chemistry Chemical Physics. 2019;21(19):10163–70.
80. Santana-Romo F, Duarte Y, Castillo F, Maestro MA, Zacconi FC. Microwave-Mediated Synthesis of N-allyl/Propargyl Derivatives: Enzymatic Analysis as a Potential Factor Xa (FXa) Inhibitor, Theoretical and Computational Molecular Docking. International Journal of Chemical Engineering and Applications [Internet]. 2020 Feb;11(1):34–41. Available from: http://www.ijcea.org/index.php?m=content&c=index&a=show&catid=109&id=1182
81. Santana-Romo F, Lagos CF, Duarte Y, Castillo F, Moglie Y, Maestro MA, et al. Innovative three-step microwave-promoted synthesis of N-propargyltetrahydroquinoline and 1,2,3-triazole derivatives as a potential factor Xa (FXA) inhibitors: Drug design, synthesis, and biological evaluation. Molecules. 2020;25(3).
82. Dodson E. The befores and afters of molecular replacement. Acta Crystallogr D Biol Crystallogr. 2008 Jan 1;64(1):17–24.
83. García-González G, González GM, Palma-Nicolás JP. Las proteasas de serina bacterianas y su implicación en la fisiopatología de la infección. Revista del Laboratorio Clínico. 2019 Jul;12(3):137–46.
84. Duarte JM, Dutta S, Goodsell DS, Burley SK. Exploring protein symmetry at the RCSB Protein Data Bank. Emerg Top Life Sci. 2022 Sep 9;6(3):231–43.
85. Fan J, Fu A, Zhang L. Progress in molecular docking. Quantitative Biology. 2019 Jun 4;7(2):83–9.
86. Brandenburg F, Klähn S. Small but Smart: On the Diverse Role of Small Proteins in the Regulation of Cyanobacterial Metabolism. Life. 2020 Dec 1;10(12):322.
87. Abrusán G, Marsh JA. Alpha Helices Are More Robust to Mutations than Beta Strands. PLoS Comput Biol. 2016 Dec 9;12(12):e1005242.
88. Fox NK, Brenner SE, Chandonia JM. SCOPe: Structural Classification of Proteins—extended, integrating SCOP and ASTRAL data and classification of new structures. Nucleic Acids Res. 2014 Jan;42(D1):D304–9.
Received: 28 September 2023/ Accepted: 15 November 2023 / Published:15 December 2023
Citation. Santana-Romo, F.; Zacconi, F. C.; Virtual Screening and Analysis of Enzyme-Ligand Complexes for Factor Xa (FXa) in the Human Coagulation Cascade Revis Bionatura 2023;8 (4) 27. http://dx.doi.org/10.21931/RB/2023.08.04.27
Additional information Correspondence should be addressed to : [email protected]
Peer review information. Bionatura thanks anonymous reviewer(s) for their contribution to the peer review of this work using https://reviewerlocator.webofscience.com/
All articles published by Bionatura Journal are made freely and permanently accessible online immediately upon publication, without subscription charges or registration barriers.
Bionatura ISSN. 13909355. Scopus coverage years: from 2016 to the Present
Publisher's Note: Bionatura stays neutral concerning jurisdictional claims in published maps and institutional affiliations.
Copyright: © 2023 by the authors. They were submitted for possible open-access publication under the terms and conditions of the Creative Commons Attribution (CC BY) license (https://creativecommons.org/licenses/by/4.0/).
