2024..01.01.30
Files > Conference Series > 2024 > Chimborazo
Protocolo sencillo y eficiente para la extracción de ADN a partir de semillas de cafeto (Coffea canephora Pierre)
Simple and efficient protocol for DNA extraction from the coffee tree (Coffea canephora Pierre) seeds
Yanelis Castilla Valdés1*, Natacha Soto Pérez2, María Esther González Vega3
1Instituto Nacional de Ciencias Agrícolas, Mayabeque, Cuba.
2Centro de Ingeniería Genética y Biotecnología, Habana, Cuba.
3Instituto Nacional de Ciencias Agrícolas, Mayabeque, Cuba.
*Correspondencia: [email protected]
Available from. http://dx.doi.org/10.21931/BJ/2024.01.01.30
RESUMEN
El primer paso para realizar cualquier tipo de estudio genético es contar con un protocolo para el aislamiento de ADN. Aunque el material vegetal más utilizado para ello es el tejido foliar, cuando no existe disponibilidad, las semillas constituyen otra opción y además, durante su traslado del campo al laboratorio no se produce degradación del ADN ni se necesita nitrógeno líquido para su conservación. Sin embargo, la extracción de ADN a partir de semillas de algunas especies como el cafeto, resulta complicado debido a su dureza y contenido de compuestos fitoquímicos. En este trabajo nos propusimos optimizar un protocolo de extracción de ADN de semillas de cafeto (C. canephora) para ser utilizado en análisis moleculares. Se utilizó el método CTAB como control y tres variantes de este. La calidad del ADN se comprobó en geles de agarosa y su concentración fue determinada en un nano-espectrofotómetro. Se realizó la amplificación del ADN por PCR con un cebador de RAPD, y su chequeo en electroforesis. Se logró obtener ADN libre de grasas y polisacáridos, con suficiente calidad para ser utilizado en PCR, como se demostró por la observación en gel, los valores de concentración y relaciones de absorbancia, y la obtención de las bandas que produjo el cebador. Este protocolo permite el acceso a la caracterización genética del cafeto, esencial para la conservación de su diversidad y para el mejoramiento genético.
Keywords: ADN, cafeto, extracción, genética, mejoramiento, semillas.
ABSTRACT
The first step in any genetic study is to have a protocol for DNA isolation. Although leaf tissue is the most commonly used plant material for this purpose, seeds are another option when leaf tissue is not available. In addition, during seed transfer from the field to the laboratory, DNA degradation does not occur, and liquid nitrogen is not needed for preservation. However, DNA extraction from seeds of some species, such as coffee trees, is complicated due to their hardness and content of phytochemical compounds. In this work, we propose optimizing a DNA extraction protocol from coffee tree (C. canephora) seeds for molecular analysis. The CTAB method was used as a control and three variants of this method. DNA quality was checked in agarose gels, and its concentration was determined using a nano-spectrophotometer. DNA amplification was performed by PCR with a RAPD primer and checked in electrophoresis. It was possible to obtain DNA free of fats and polysaccharides, with sufficient quality for PCR. The quality was demonstrated by gel observation, concentration values, and absorbance ratios, as well as by obtaining the bands produced by the primer. This protocol allows access to the genetic characterization of the coffee plant, essential for conserving its diversity and for genetic improvement.
Keywords: DNA, coffee tree, extraction, genetics, breeding, seeds.
INTRODUCCIÓN
El cafeto (Coffea spp.) representa un importante papel en la economía global, pues se considera una de las principales fuentes de ingreso en unos 80 países que producen café1. Aunque se conocen 130 especies de Coffea spp, originarias de África tropical, las islas del Océano Índico, Asia y Australia2, las dos principales especies cultivadas son Coffea arabica L. (café Arábica) y C. canephora Pierre (café Robusta)3.
En el siglo XVIII fueron introducidas las primeras plantas de cafeto en Latinoamérica, donde se produce en la actualidad el 80% del volumen de exportación mundial de café4. En el año 2022 su comercio global abarcó unos 171 268 000 sacos. Cuba se encuentra en el séptimo lugar entre los países productores de café en el área del Caribe, América Central y México, y se estima que su producción creció en un 0,7% en el período entre 2020 y 20225. En Cuba, el café es una bebida muy demandada por la población, por lo que constituye una prioridad en el sector de la agricultura y un renglón exportable6.
A nivel global se ha observado una afectación de la producción de café por factores abióticos como el incremento de las temperaturas y la disminución de las lluvias, asociados al cambio climático. Asimismo, la influencia de estos elementos abióticos, puede incrementar el impacto de factores bióticos como las plagas y enfermedades3. La erosión genética in situ de las especies de cafeto también resulta preocupante, con aproximadamente el 60% de ellas enfrentando alguna forma de amenaza en la vida silvestre y con poca representación en colecciones ex situ7.
El acceso a una efectiva caracterización genética resulta esencial para la conservación de la diversidad genética y para el exitoso mejoramiento genético de los cultivares3. En general, los programas de mejora genética de frutales tropicales y subtropicales resultan poco eficaces y costosos. Para aumentar su eficacia, se necesitan métodos moleculares sencillos, económicos y fiables para la caracterización del germoplasma disponible, como la aplicación de marcadores de ADN (Ácido desoxirribonucleico), los cuales pueden contribuir a acelerar los programas de mejoramiento. En especies de frutales arbóreos tropicales, resulta un reto la extracción de ADN genómico de calidad, para ser utilizado en las técnicas de marcadores moleculares basados en PCR (Reacción en Cadena de la Polimerasa). Esto se debe a la presencia de contaminantes que pueden dificultar la aplicación de métodos moleculares al purificarse conjuntamente con el ADN durante las extracciones8.
Las especies de cafeto poseen abundantes sustancias fitoquímicas en órganos como las hojas y los frutos. Entre ellas, se encuentran compuestos fenólicos, alcaloides, carotenoides, terpenoides, sustancias inorgánicas y vitaminas, entre otros9. Sus semillas son ricas en lípidos, con 15% en C. arabica y aproximadamente un 10% en C. canephora var. Robusta10. Estos lípidos se localizan en “cuerpos grasos” situados en el endospermo11.
Aunque generalmente para el aislamiento de ADN para análisis moleculares se utilizan muestras foliares, en ocasiones no se cuenta con disponibilidad de ellas y las semillas resultan el material más accesible, y también fácil de transportar de un lugar a otro12.
Se han descrito diferentes protocolos para la extracción de ADN de cafeto a partir del tejido foliar13-17. Para la extracción de ADN a partir de semillas, hay pocos protocolos y la mayoría de ellos fueron establecidos en C. arabica3, 18-20. La composición química del material vegetal varía de una especie o variedad de cafeto a otra, lo cual influye sobre la efectividad del método de extracción15. Un mismo método de extracción pudiera no proporcionar los mejores rendimientos de ADN en diferentes genotipos, por lo que es esencial un aislamiento eficaz del ADN y la optimización de la PCR. Para obtener ADN de la calidad adecuada, los investigadores suelen modificar una técnica o combinar dos o más procesos distintos17. Por todo ello, en el presente trabajo nos propusimos optimizar un protocolo de extracción de ADN de semillas de cafeto (C. canephora) con la calidad necesaria para ser utilizado en análisis moleculares.
MATERIALES Y MÉTODOS
Material vegetal
Se utilizaron semillas de cafeto (Coffea canephora Pierre), variedad Robusta, que forman parte de una colección de trabajo del INCA (Instituto Nacional de Ciencias Agrícolas, Cuba). Se escogió el genotipo M-28 (Figura 1A, B), por su resistencia o tolerancia a estrés hídrico y a algunas plagas que afectan al cultivo21.

Figura 1: Planta y semillas de cafeto (Coffea canephora Pierre), variedad Robusta.
Extracción de ADN
Previamente se utilizó el protocolo CTAB22 para la extracción del ADN total de semillas, pero la calidad y concentración fue muy baja por el elevado contenido de grasas. Por ello, se hicieron modificaciones al protocolo CTAB y se evaluaron tres variantes con cuatro réplicas (grupos de semillas) cada una. Se maceraron las semillas en morteros y se utilizaron 100 mg/réplica. El protocolo CTAB se usó como control.
Variante 1: Las semillas se maceraron y el material macerado se lavó por tres veces con 3 mL de éter de petróleo, hasta eliminar la grasa. Después se procedió con el protocolo CTAB.
Variante 2: En el paso de precipitación con isopropanol frío, se añadió una solución de cloruro de sodio (5 M), se mezcló y se dejó precipitar a -20 °C por 16 h12.
Variante 3: Aquí se combinaron las variantes 1 y 2. El lavado con éter de petróleo después de la maceración y la adición de cloruro de sodio (5 M) en la precipitación con isopropanol a -20 °C por 16 h.
La calidad del ADN se visualizó en gel de agarosa al 0,8% con bromuro de etidio. La calidad y concentración del ADN también se analizó en un nano-espectrofotómetro (NP80, IMPEM, Alemania).
Amplificación del ADN por PCR y chequeo en electroforesis
La calidad del ADN se confirmó mediante PCR con el cebador OPC 04 (CCG CAT CTA C) para RAPD (Random Amplified Polimorfic DNA, por sus siglas en inglés), que ha sido utilizado en cafeto23. La mezcla para la reacción de PCR contenía ADN genómico (50 ng), tampón Go Taq Green 5x (Promega) (10 mM), dNTP mix (Promega) (10 mM), cebador (20 pmol/µL) y Taq ADN Polimerasa (Promega) (5 U), en un volumen final de 25 µL. Para la amplificación del ADN se utilizó un termociclador Gene Touch Bioer con el siguiente programa: un ciclo inicial de 95 °C por 3 min; 40 ciclos de 94 °C por 1 min, 38 °C por 1 min y 72 °C por 2 min y un ciclo final de 72 °C por 5 min.
El producto de la amplificación del PCR se visualizó en gel de agarosa al 1%. Se utilizó el marcador de peso molecular 1 kb (Promega). El PCR se repitió dos veces para confirmar el patrón específico.
RESULTADOS Y DISCUSIÓN
Obtención de un ADN de calidad
La extracción de ADN a partir de semillas de cafeto, es un proceso laborioso en comparación con otras especies forestales, ya que además de la dureza de las semillas, estas contienen elevadas cantidades de polisacáridos, polifenoles, lípidos, y varios metabolitos secundarios como alcaloides, flavonoides y fenoles, que interfieren durante el aislamiento de este ácido nucleico. Las diferentes variantes de extracción de ADN estudiadas, mostró un protocolo que permite obtener ADN con la calidad necesaria para ser amplificado por PCR (Figura 2).
Figura 2: ADN de semillas de cafeto (C. canephora Pierre) visualizado en gel de agarosa. Leyenda: A: Protocolo CTAB (control). B: Variante 1. C: Variante 2. D: Variante 3. Pocillos: réplicas del ADN extraído en cada variante.
En el protocolo CTAB usado como control, se obtuvieron bajos valores de concentración de ADN a partir de las semillas, como se muestra en la tabla 1.
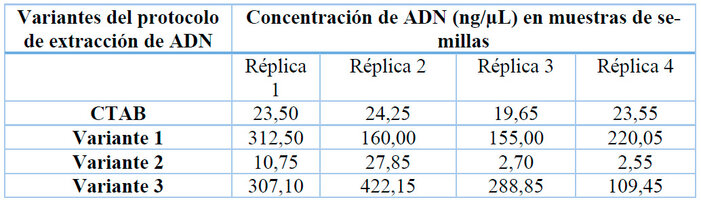
Tabla 1: Concentración de ADN (ng/µL) extraído de semillas de cafeto (C. canephora Pierre).
Esto pudiera deberse al elevado contenido de grasas presentes en el material macerado, característico de las semillas de cafeto10, lo cual limitó la obtención de una adecuada cantidad y calidad de ADN (Figura 2 A, B, C).
En la variante 1 (Figura 2 B), las concentraciones de ADN estuvieron por debajo de 312,50 ng/µL (Tabla 1) y los mayores valores de las relaciones de la absorbancia, indicativas de la calidad del mismo (A260/A280 y A260/A230), fueron de 1,31 y 0,47; respectivamente (Tabla 2). El ADN no tuvo la calidad necesaria para realizar el PCR, ya que, para una calidad óptima, los resultados de estas relaciones deben encontrarse entre 1,8 y 212. En esta variante se observó un menor contenido de grasas en las muestras, posiblemente por el efecto de la utilización del éter de petróleo en la maceración de las semillas.
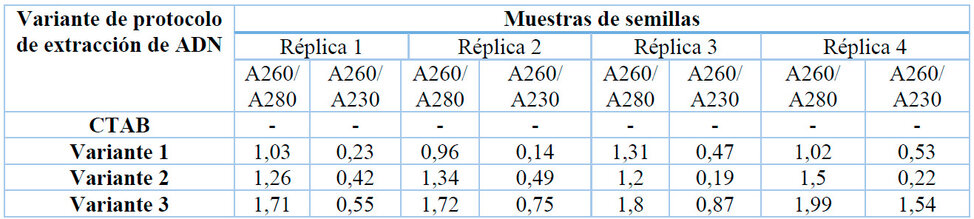
Tabla 2: Evaluación de la calidad del ADN. Relaciones de absorbancia en el ADN extraído de semillas de cafeto (C. canephora Pierre).
Hasta el momento, no se tiene conocimiento de que el éter de petróleo haya sido utilizado para la eliminación de grasas, en otros protocolos de extracción de ADN, a partir de semillas de cafeto, sin embargo, sí se ha empleado como solvente para la determinación del contenido de lípidos en granos molidos de cafeto24. Las semillas de otras especies como el cacao (Theobroma cacao) y el cacao blanco (Theobroma grandiflorum) también son ricas en carbohidratos, proteínas, compuestos fenólicos y grasas. En estas semillas se utilizó el éter de petróleo para la remoción de grasas, previo a la utilización de un protocolo para la extracción de ADN, con resultados satisfactorios25.
En la variante 2, las concentraciones de ADN variaron entre 2,55 y 27,85 ng/µL (Tabla 1), y la relación A260/A280 fue de 1,50 como máximo, así como la de A260/A230 fue de 0,49 como máximo (Tabla 2). Se visualizó muy poco ADN en el gel de agarosa (Figura 2 C), por lo que la concentración del ADN obtenido se considera baja, y su calidad aún no se considera óptima. En esta variante se utilizó cloruro de sodio a altas concentraciones, para contribuir a la eliminación de polisacáridos26 que abundan en las semillas de cafeto9. Sin embargo, su empleo no fue suficiente para eliminar los contaminantes que afectaban las muestras, por lo que en la variante 3 se combinó con la utilización del éter de petróleo.
Algunos autores refieren que el cloruro de sodio favorece la eliminación de las proteínas que están unidas al ADN. También ayuda a mantener las proteínas disueltas en la capa acuosa para que no precipiten en el alcohol junto con el ADN, neutralizando así las cargas negativas del ADN para que las moléculas puedan unirse27. Las concentraciones elevadas de cloruro de sodio (5 M) se utilizan para prevenir la contaminación de la muestra con polisacáridos que afectan la pureza del ADN. Estos pueden inhibir la actividad de enzimas como polimerasas, ligasas y endonucleasas de restricción. La base para separar los polisacáridos de los ácidos nucleicos, es su solubilidad diferencial en presencia de las altas concentraciones de NaCl, ya que los polisacáridos precipitan bajo la acción de fuerzas centrífugas26.
En la variante 3, se obtuvieron los valores más elevados de concentraciones de ADN (Tabla 1), y la relación A260/A280 máxima alcanzada fue de 1,99; mientras que la de A260/A230 fue de 1,54 (Tabla 2). En esta variante se obtuvo una mayor cantidad y calidad de ADN (Figura 2 D), lo cual posibilitó la realización del PCR. Las modificaciones que se le realizaron al protocolo original permitieron eliminar algunas de las sustancias fuentes de contaminación e impurezas, como polisacáridos y grasas, que afectan la extracción de ADN. En consecuencia, esta fue la variante de extracción de ADN utilizada para la amplificación del ADN por PCR.
Eficiente amplificación del ADN mediante PCR
La amplificación del ADN de las semillas de cafeto utilizando un cebador RAPD, demostró la eficiencia y confiabilidad de la tercera variante del método de extracción de ADN evaluado (Figura 3).
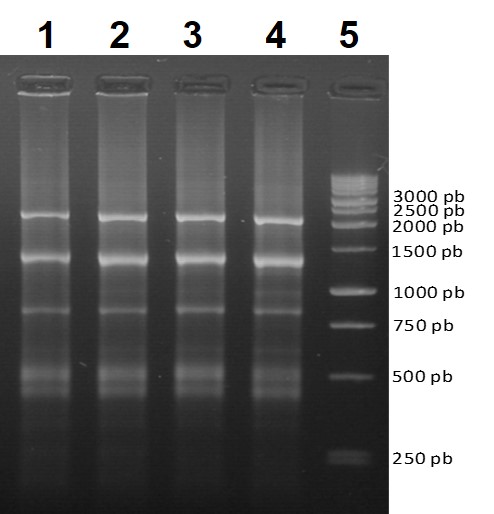
Figura 3: Patrones de bandas RAPD con el cebador OPC 04 en semillas de cafeto (C. canephora). Pocillos 1- 4: Bandas amplificadas del PCR con ADN de la variante 3; Pocillo 5: Marcador de peso molecular 1Kb (Promega).
El cebador OPC 04 produjo bandas visibles, bien definidas y reproducibles. Se originaron un total de cinco bandas y no se encontraron bandas polimórficas al comparar los patrones obtenidos a partir de los diferentes grupos de semillas.
Algunos investigadores20 utilizaron un protocolo CTAB modificado, para la extracción de ADN de semillas de cafeto y otras especies. Estas modificaciones se basaron en añadir PVP (polivinilpirrolidona) y carbón activado al buffer de extracción antes de la incubación de la muestra. Según los autores, los compuestos liberados por el citosol deben entrar en contacto con el carbón activado antes que el ADN, y el PVP propicia de manera sinérgica, la unión de los polifenoles al carbón activado. De esa forma, aumentan en gran medida las posibilidades de amplificación del ADN al evitar sus interacciones irreversibles con polifenoles. Este protocolo20, aunque se considera relativamente sencillo, no permitió obtener un elevado rendimiento de ADN, necesario para una adecuada reacción de PCR, tanto en cafeto como en otras especies estudiadas.
Se han probado otros protocolos donde añadieron PVP y carbón activado a los granos verdes de cafeto antes de molerlos19. Estos autores utilizaron un procedimiento CTAB modificado, con la adición de la sal acetato de sodio en alta concentración (5M) para la eliminación de polisacáridos, sin embargo, en la etapa final se requirió de un paso adicional de centrifugación durante 10 min ya que se observaron muestras con rastros residuales de polisacáridos.
Por otra parte, la metodología desarrollada por investigadores de la India12 resultó eficiente para la extracción de ADN a partir de semillas de cafeto, en cambio, incluye una serie de pasos adicionales de lavado del material. En contraste, nuestro protocolo es más simple ya que sólo modifica al CTAB en el lavado inicial con éter de petróleo (en lugar de utilizar Nitrógeno líquido, que puede resultar un recurso limitante para algunos laboratorios) y la inclusión de NaCl al isopropanol frío, por lo que se considera que resulta una alternativa eficiente para este propósito, teniendo en cuenta los valores de concentración y calidad del ADN obtenido, así como el éxito en la amplificación del ADN por PCR.
CONCLUSIONES
En este trabajo se logró optimizar un protocolo de extracción de ADN a partir de semillas de cafeto, con la calidad necesaria para ser utilizado en análisis moleculares. Este método también puede resultar útil para otros estudios de diagnóstico molecular en esta importante especie cultivada y podría extenderse a las semillas de especies de plantas que poseen abundantes compuestos fitoquímicos en estos órganos.
Contribuciones de los autores: Conceptualización, Y.C.V. y N.S.P.; metodología, Y.C.V. y N.S.P.; recursos, N.S.P. y M.E.G.V.; investigación, Y.C.V. y N.S.P.; escritura-preparación del manuscrito original, Y.C.V.; escritura-revisión y edición, N.S.P. y M.E.G.V.
Agradecimientos: Las autoras desean reconocer a los integrantes del grupo de Biotecnología de la Soya, del Departamento de Biotecnología de las Plantas, del Centro de Ingeniería Genética y Biotecnología, por su valiosa contribución al desarrollo de este estudio.
Conflictos de interés: Las autoras declaran no tener conflictos de interés.
REFERENCIAS
1. Bramel P, Krishnan S, Horna D, Lainoff B, Montagnon C. Global conservation strategy for coffee genetic resources [Internet]. Crop Trust- World Coffee Research; 2017 [Consultado 24 Sept 2023]. 71 p. Disponible en: https://worldcoffeeresearch.org/media/documents/Coffee_Strategy_Low_Res.pdf.
2. Davis AP, Rakotonasolo F. Six new species of coffee (Coffea) from northern Madagascar. Kew Bull. 2021; 76: 497-511.
3. Krishnan S, Matsumoto T, Nagai C, Falconer J, Shriner S, Long J, Medrano JF, Vega F E. Vulnerability of coffee (Coffea spp.) genetic resources in the United States. Genet Resour Crop Evol. 2021; 68: 2691-710.
4. Aguilar ME, Ortiz JL, Mesén F, Jiménez D, Altmann F. Cafe Arabica Coffea arabica L. En Jain SM, Gupta PPK, editores. Step Wise Protocols for Somatic Embryogenesis of Important Woody Plants [Internet]. Springer Nature; 2018 [Consultado 18 Sept 2023]. p. 39-62. Disponible en: https://link.springer.com/chapter/10.1007/978-3-319-79087-9_3.
5. International Coffee Organization. Coffee Report and Outlook [Internet]. 2023 [Consultado 11 Oct 2023]. Disponible en: https://icocoffee.org/documents/cy2022-23/Coffee_Report_and_Outlook_April_2023_-_ICO.pdf.
6. González ME, Rosales PR, Castilla Y, Lacerra JA, Ferrer M. Efecto del Bioenraiz® como estimulante de la germinación y el desarrollo de plántulas de cafeto (Coffea arabica L.). Cultivos Tropicales. 2015; 36(1): 73-9.
7. Davis AP, Chadburn H, Moat J, O’Sullivan R, Hargreaves S, Lughadha EN. High extinction risk of wild coffee species and implications for coffee sector sustainability. Sci Adv [Internet]. 2019 [Consultado 31 Oct 2023]; 5(1): eaav3473. Disponible en: https://doi.org/10.1126/sciadv.aav3473.
8. Ramírez IM, Rodríguez NN, Valdés-Infante J, Capote M, Becker D, Rohde W. Isolation of genomic DNAs from the tropical fruit trees avocado, coconut, guava and mango for PCR-based DNA marker application. Cultivos Tropicales. 2004; 25(1): 33-8.
9. Patay ÉB, Sali N, Kőszegi T, Csepregi R, Balázs VL, Németh TS, Németh T, Papp N. Antioxidant potential, tannin and polyphenol contents of seed and pericarp of three Coffea species. Asian Pac J Trop Med. 2016; 9(4): 366-71.
10. Speer K, Kölling-Speer I. The lipid fraction of the coffee bean. Braz J Plant Physiol. 2006; 18(1): 201-16.
11. Antoine G, Vaissayre V, Meile JC, Payet J, Conéjéro G, Costet L, Fock-Bastide I, Joët T, Dussert S. Diterpenes of Coffea seeds confer defense against fungi and insects and are transferred from the endosperm to the seedling after germination. Plant Physiol Biochem. 2023; 194: 627-37.
12. Kumar M, Sandhya N, Ram AS, Sreenath HL, Jayarama. A simple method of DNA extraction from coffee seeds suitable for PCR analysis. Afr J Biotechnol. 2008; 7(4): 409-13.
13. Diniz LEC, Ruas CF, Carvalho VP, Torres FM, Ruas EA, Santos MO, Sera T, Ruas PM. Genetic diversity among forty coffee varieties assessed by RAPD markers associated with restriction digestion. Braz Arch Biol Technol. 2005; 48(4): 511-21.
14. Bressan EA, Rossi ML, Gerald LT, Figueira A. Extraction of high-quality DNA from ethanol-preserved tropical plant tissues. BMC Res Notes [Internet]. 2014 [Consultado 31 Oct 2023]; 7: 268. Disponible en: http://www.biomedcentral.com/1756-0500/7/268.
15. Suazo T, Miranda S, Rivers E, Lacayo M, Tenorio D. Evaluación de metodologías de extracción de ADN de plantas recalcitrantes. Revista Torreón Universitario. 2020; 9(24): 45-57.
16. Parveen A, Kumar D. Comparative study of different approaches to isolate DNA from Coffea arabica leaves. Glob J Res Anal. 2023; 12(5): 52-3.
17. Pokharel S, Khanal BC, Basnetb A, Pandey GR, Basnet S. DNA extraction and PCR optimization for DNA barcode analysis of commercially-grown coffee varieties in Nepal. J Sci Eng Technol [Internet]. 2023 [Consultado 30 Nov 2023]; 17(1). Disponible en: https://journals.ku.edu.np/kuset/article/view/124/48.
18. Crochemore M L, Nunes ML, Andrade GA, Molinare HBC, Vasconcellos ME. Varietal identification of coffee seeds by RAPD technique. Braz Arch Biol Technol. 2004; 47(1): 7-11.
19. Martellossi C, Taylor EJ, Lee, D, Graziosi G, Donini P. DNA Extraction and analysis from processed coffee beans. J Agric Food Chem. 2005; 53(22): 8432-436.
20. Križman M, Jakše J, Baričevič D, Javornik B. Robust CTAB-activated charcoal protocol for plant DNA extraction. Acta Agric Slov. 2006; 87(2): 427-33.
21. González ME, Rodríguez J. Respuesta de plantas de Coffea canephora a la inoculación con hongos micorrizógenos arbusculares durante la fase de aclimatización. Cultivos Tropicales. 2004, 25(1): 13-6.
22. Doyle JJ, Doyle JL. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin. 1987; 19(1): 11-5.
23. Vargas-Segura C, López-Gamboa E, Araya-Valverde E, Valdez-Melara M, Gatica-Arias A. Sensitivity of seeds to chemical mutagens, detection of DNA polymorphisms and agro-metrical traits in M1 generation of coffee (Coffea arabica L.). J Crop Sci Biotechnol. 2019; 22: 451-64.
24. Sant’Ana GC, Pereira LFP, Pot D, Ivamoto ST, Domingues DS, Ferreira RV, Pagiatto N F, da Silva BSR, Nogueira LM, Kitzberger CSG, Scholz MBS, de Oliveira FF, Sera GH, Padilha L, Labouisse J-P, Guyot R, Charmetant P, Leroy T. Genome-wide association study reveals candidate genes influencing lipids and diterpenes contents in Coffea arabica L. Sci Rep. [Internet]. 2018 [Consultado 30 Nov 2023]; 8, 465. Disponible en: https://doi.org/10.1038/s41598-017-18800-1
25. Ramos SNM, Salazar MM, Pereira GAG, Efraim P. Plant and metagenomic DNA extraction of mucilaginous seeds. MethodsX. 2014; 1: 225-28.
26. Velasco R. Marcadores moleculares y la extracción de ADN. Biotecnología en el Sector Agropecuario y Agroindustrial. 2005; 3(1): 14-8.
27. Heikrujam J, Kishor R, Mazumder PB. The chemistry behind plant DNA isolation protocols. En: Boldura OM, Baltă C, Awwad NS, editores. Biochemical. Analysis Tools - Methods for Bio-Molecules Studies [Internet]. IntechOpen; 2020 [Consultado 16 Sep 2023]. Disponible en: http://dx.doi.org/10.5772/intechopen.92206.
Received: 9 October 2023/ Accepted: 15 January 2024 / Published: 15 February 2024
Citation: Castilla-Valdés Y., Soto-Pérez N., González-Vega M. E. Simple and efficient protocol for DNA extraction from coffee tree (Coffea canephora Pierre) seeds. Bionatura Journal 2024; 1 (1) 30. http://dx.doi.org/10.21931/BJ/2024.01.01.30
Additional information Correspondence should be addressed to [email protected]
Peer review information. Bionatura Journal thanks anonymous reviewer(s) for their contribution to the peer review of this work using https://reviewerlocator.webofscience.com/
All articles published by Bionatura Journal are made freely and permanently accessible online immediately upon publication, without subscription charges or registration barriers.
Publisher's Note: Bionatura Journal stays neutral concerning jurisdictional claims in published maps and institutional affiliations.
Copyright: © 2024 by the authors. They were submitted for possible open-access publication under the terms and conditions of the Creative Commons Attribution (CC BY) license (https://creativecommons.org/licenses/by/4.0/).
