2024..01.01.74
Files > Conference Series > 2024 > Chimboazo ild pagina nueva
In silico design of CRISPR/Cas9 guide RNA for the knockout of the phytoene desaturase gene in sweet potato (Ipomoea batatas L.)
Diseño in silico de ARN guía CRISPR/Cas9 para la inactivación del gen fitoeno desaturasa en camote (Ipomoea batatas L.)
Mao Yupanqui-Celestino 1, Brayan Lugo 1, Giselle Jazmin Aguilar-Balabarca 1, Allison Esmeralda Lozano-Galindo 1, Desiderio Elías Cotos-Durán 1, Carlos Roberto Pesantes-Rojas 2, Miguel Angel Aguilar-Luna-Victoria 3, Ernesto Díaz-Ronceros 4, Linder Ramírez-Viena 5,*
1Escuela Profesional de Biología con mención en Biotecnología, Facultad de Ciencias, Universidad Nacional José Faustino Sánchez Carrión, Huacho, Perú.
2Escuela Profesional de Matemática Aplicada, Facultad de Ciencias, Universidad Nacional José Faustino Sánchez Carrión, Huacho, Perú.
3Escuela Profesional de Estadística e Informática, Facultad de Ciencias, Universidad Nacional José Faustino Sánchez Carrión, Huacho, Perú.
4Escuela Profesional de Ingeniería Electrónica, Facultad de Ingeniería Industrial, Sistemas e Informática, Universidad Nacional José Faustino Sánchez Carrión, Huacho, Perú.
5Departamento de Ciencias Naturales, Facultad de Educación, Universidad Nacional José Faustino Sánchez Carrión, Huacho, Perú
* Correspondence: Linder Ramírez-Viena ([email protected]).
Available from. http://dx.doi.org/10.21931/BJ/2024.01.01.74
ABSTRACT
This study aimed to design in silico guide RNA (sgRNA) for CRISPR/Cas9-mediated knockout of the phytoene desaturase (PDS) gene in sweet potato (Ipomoea batatas L.). The sequence of the coding region of the IbPDS gene is 1791 base pairs (bp) long, and these, in turn, are equivalent to 572 amino acids. The amino acid sequence of the IbPDS gene was compared with the homologous sequences of other nearby plant species, showing that it presents a close similarity with PDS of Ipomoea triloba and Ipomoea nil with 98.60% and 97.73%, respectively. CRISPR RGEN Tools provided 113 results for the IbPDS gene, filtering to 24 and selecting three sgRNA sequences for the design of the gene editing vector, which were sgRNA 1 (5'-ACCTCATCAGTCACCCTGTCNGG-3'), sgRNA 2 (5'- CCTCCAGCAGCAGTATTGGTTGGTTTGNGG -3') and sgRNA 3 (5'- CTGAACTCTCCTGGTTGGTTGTTNGG -3'). The predicted secondary structures of the selected sgRNAs present efficient sgRNA structures for gene editing of the target gene. The PMH-Cas9-3xsgRNA vector for CRISPR/Cas9-mediated knockout of the IbPDS gene was designed in silico with three sgRNA sequences and one Hygromycin resistance marker.
Keywords: Gene editing, sgRNA, IbPDS, gene editing vector, Hygromycin.
RESUMEN
Este estudio tuvo como objetivo el diseñó in silico de ARN guía (sgRNA) para la inactivación del gen fitoeno desaturasa (PDS) mediada por CRISPR/Cas9 en camote (Ipomoea batatas L.). La secuencia de la región codificante del gen IbPDS presenta una longitud de 1791 pares de base (bp) y estos a su vez equivalen a 572 aminoácidos. Se comparó la secuencia de aminoácidos del gen IbPDS con las secuencias homólogas de otras especies vegetales cercanas, demostrándose que presenta una similitud cercana con PDS de Ipomoea triloba e Ipomoea nil con 98.60% y 97.73%, respectivamente. CRISPR RGEN Tools proporcionó 113 resultados de sgRNA para el gen IbPDS, filtrando a 24 secuencias y seleccionando tres secuencias de sgRNA para el diseño del vector de edición genética, los cuales fueron: sgRNA 1 (5’-ACCTCATCAGTCACCCTGTCNGG-3’), sgRNA 2 (5’- CCTCCAGCAGTATTGGTTTGNGG -3’) y sgRNA 3 (5’- CTGAACTCTCCTGGTTTGTTNGG -3’). La predicción de las estructuras secundarias de los sgRNA seleccionados presentan estructuras de sgRNA eficientes para la edición genética del gen diana. Se diseño in silico el vector pMH-Cas9-3xsgRNA, vector para la inactivación del gen IbPDS mediada por CRISPR/Cas9 con tres secuencias de sgRNA y un marcador de resistencia a Higromicina.
Palabras claves: Edición genética, sgRNA, IbPDS, vector de edición genética, Higromicina.
INTRODUCCIÓN
La edición genética es una de las tecnologías más importantes para la investigación moderna. Esta implica nucleasas específicas para generar mutaciones en regiones del genoma de organismos. Así mismo, ha aumentado su desarrollo en la edición genómica basada en CRISPR (Clustered Regulary Interspaced Short Palindromic Repeats) /Cas9 (CRISPR Associated Protein 9) que ha revolucionado por completo la biotecnología1. Actualmente, es usada para crear mutaciones dirigidas en el genoma del organismo de interés con resultados de amplia importancia en la biología funcional.
El sistema CRISPR/Cas9 de edición genética es capaz de cortar ADN cromosómico en regiones específicas. Posterior al corte, la célula utiliza mecanismos de reparación en el corte del ADN. Estas nucleasas inducen una rotura de doble cadena (DSB) en la secuencia del gen. El DSB se repara mediante mecanismos de recombinación homóloga (HDR) o de unión no homóloga propenso a errores (NHEJ), siendo este último el más predominante, debido a que genera pérdida de información genética a través de inserciones y deleciones (indels). Si esta se produce en regiones codificantes de un gen puede resultar en pérdida de su funcionalidad2.
CRISPR/Cas9 tiene la capacidad de generar la pérdida o ganancia de funciones de genes, expresando características de interés en plantas3. Actualmente la técnica de inactivación de genes mediante CRISPR/Cas9 es la más usada por su fácil manipulación y alta precisión4. El mejoramiento de plantas mediado por la edición genética ha permitido añadir características genéticas de interés industrial en cultivos como especies oleaginosas ricas en ácidos grasos omega 3 y el arroz de alto rendimiento5.
Los de ARN guías (sgRNA) permiten dirigir a la enzima Cas9 a regiones específicas del ADN, estas secuencias blanco serán cortadas de manera específica. Así el diseño in silico de sgRNA garantizará la síntesis correcta de una nueva molécula de ARN para redireccionar a la nucleasa hacia diferentes regiones del gen de interés. En ese sentido, CRISPR se distingue de las nucleasas porque no requiere diseñar una nueva enzima por cada secuencia que se desee editar con precisión. De manera que se obtiene una alta probabilidad de variaciones genéticas que puedan alterar a genes funcionales6.
El camote (Ipomoea batatas L.) es una de las especies más cultivadas en Perú, lo que ha posicionado al país como el cuarto mayor exportador a nivel mundial. La concentración alta de almidón en sus raíces hace de estas una de las principales fuentes de nutrición humana7. El cloroplasto es la organela que lleva a cabo la síntesis de almidón por medio de cinco enzimas centrales, además, de codificar los carotenos de pigmentación, convirtiendo al genoma plastídico en un indicador cualitativo óptimo para pruebas de edición genética8.
El gen fitoeno desaturasa (PDS) tiene como principal objetivo la biosíntesis de carotenoides y su función compromete muchos rasgos fenotípicos característicos como el color. Incluso, la secuencia de la proteína PDS se ha mantenido evolutivamente constante a lo largo del tiempo, lo que sugiere que este gen, en específico, es de gran aporte para los métodos de edición genética9. Es decir, que al inducir la disfuncionalidad del gen PDS permitirá determinar de forma rápida el grado de éxito en la edición genética, ya que compromete rasgos fenotípicos de gran notoriedad en la planta. Por lo tanto, el adecuado silenciamiento genético del gen PDS en Ipomoea batatas, considerada una planta de gran valor alimenticio y económico, aportará en el conocimiento global en edición genética, que es de gran interés científico, industrial y alimenticio10.
Para el desarrollo de plantas editadas genéticamente es crucial presentar protocolos de regeneración de plantas a partir de diferentes explantes iniciales mediante el uso de metodologías de cultivo de tejidos vegetales, teniendo alternativas como la regeneración mediante callos11,12, anteras13, microsporas, protoplastos, callos embriogénicos14, embriones somáticos, embriones cigóticos o directamente del explante inicial15.
La obtención de características deseadas en las plantas mejoradas a partir de las ediciones genéticas dirigidas por CRISPR/Cas9 no transfiere transgenes al individuo, por lo que los individuos obtenidos por este método no pueden ser consideradas plantas transgénicas. Como resultado obtenemos plantas de múltiples aplicaciones comerciales. La técnica de edición libre de T-DNA puede impulsar los procesos de mejoramiento genético en plantas, por ello se verifica la respuesta de especies comerciales como la Ipomoea batatas al ser editadas genéticamente16.
En este sentido, el presente estudio tiene como objetivo el diseño in silico de ARN guías específicos para la inactivación del gen fitoeno desaturasa del camote (IbPDS) mediante edición genética con el sistema CRISPR/Cas9.
MATERIALES Y MÉTODOS
Identificación de secuencias de aminoácidos del gen IbPDS
La secuencia de aminoácidos del gen fitoeno desaturasa del camote (IbPDS) estuvo basada en la secuencia presentada por Seo et al.9, y se comparó con otras secuencias de aminoácidos de PDS en otros organismos que presentaron un porcentaje de similitud superior al 80%, las secuencias se obtuvieron de NCBI (http://www.ncbi.nlm.nih.gov/). Las secuencias se alinearon utilizando el programa Bioedit y se formó un árbol filogenético mediante el método de unión de vecinos (Neighbor-Joining) con 1000 repeticiones de análisis de arranque (Bootstrap) con el programa MEGA 11.
Diseño in silico de sgRNA para el gen IbPDS
El diseño de los sgRNA para la región de destino del gen IbPDS se realizó empleando la herramienta en línea CRISPR RGEN Tools (http://www.rgenome.net/) en el genoma Ipomoea batatas Pasi3. En este proceso se empleó la función Cas-Designer con el objetivo de realizar reconocimiento y mutación de cambio de marco en la secuencia objetivo. Las secuencias objetivos aptas RGEN (5' a 3') obtenidas se evaluaron con los criterios de contenido de GC entre 40% a 60%, puntuación fuera de cuadro mayor igual a 66 y desajustes de 1-0-0. Para las secuencias seleccionadas de Cas-Designer, se realizó una búsqueda potencial fuera del objetivo con la función Cas-OFFinder para evaluar su objetivo, número de objetivos encontrados, posición, desajuste y cromosoma objetivo de las secuencias on-target y off-target, seleccionado las secuencias sgRNA que posea 0 en desajustes y 1 en número de objetivos encontrados.
Predicción in silico de la estructura de sgRNA para IbPDS
La predicción de las estructuras de los sgRNA seleccionados se realizó empleando la herramienta en línea RNAfold webserver perteneciente The ViennaRNA Web Services (http://rna.tbi.univie.ac.at/#webservices). La predicción se enfocó a las estructuras secundarias de los sgRNA seleccionados en conjunto con un RNA scaffold establecido para la metodología de edición genética mediada por CRISPR/Cas9. La selección de los sgRNA basados en sus estructuras secundarias fue establecido por la formación de dos horquillas y tres bucles, en cada extremo, superior e inferior, respectivamente.
Diseño in silico del vector de inactivación del gen IbPDS mediada por CRISPR/Cas9
Con los sgRNA seleccionados se continuó con el diseño del vector edición genética del gen IbPDS, para esto se realizó el diseño in silico con el software Snapgene. Se descargo la secuencia del vector pMH-Cas9-gate (Plasmid #113742) de la plataforma virtual de addgene (https://www.addgene.org/) (Figura 1). El vector fue modificado según lo establecido por Mallet et al.17, para la inserción de los tres sgRNA seleccionados mediante las reacciones de recombinación direccional mulisitio Gateway LR, opción integrada en el software Snapgene. Cada sgRNA previa a su inserción en el vector de destino se encuentran integrados con el promotor AtU6-26.
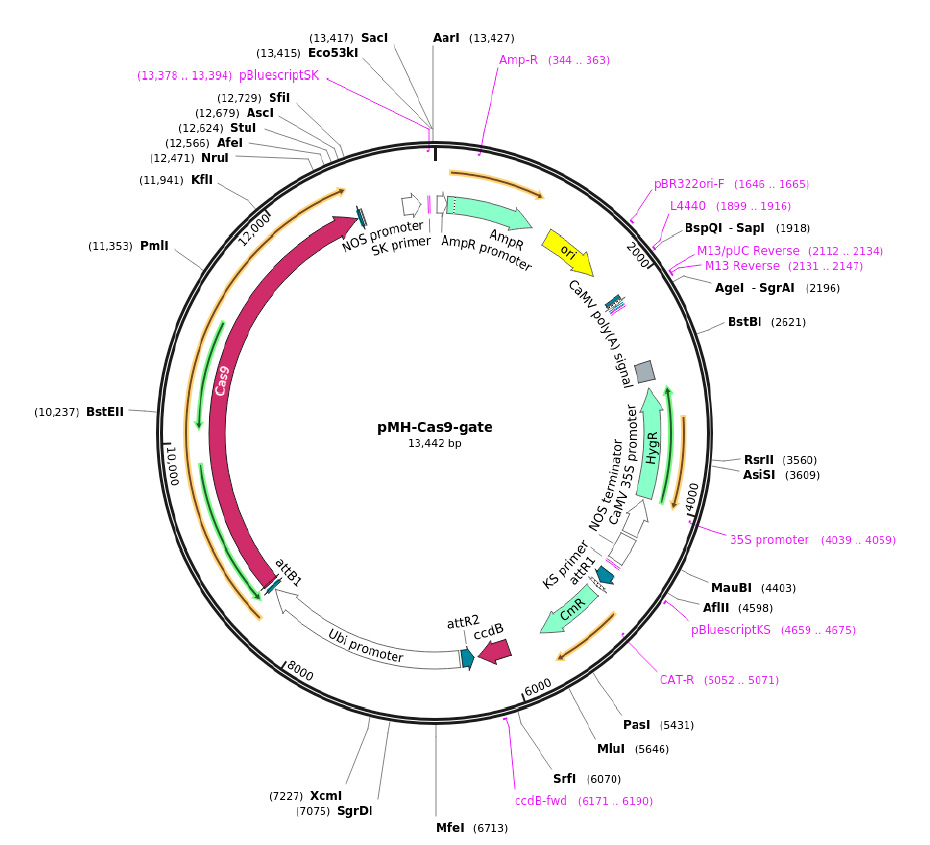
Figura 1: Mapa del vector de destino pMH-Cas9-gate que contiene los sitios Gateway.
RESULTADOS Y DISCUSIÓN
Identificación de secuencias de aminoácidos del gen IbPDS
La secuencia del fitoeno desaturasa del camote (IbPDS) presenta un tamaño total de 4741 bp, dividiéndose en 14 exones y 13 intrones, de los cuales 1791 bp representan su región codificante y estos a su vez equivalen a 572 aminoácidos, las cuales representan los datos obtenidos de Seo et al.9. La secuencia de aminoácidos de fitoeno desaturasa de camote presentó mayor porcentaje de similitud con Ipomoea triloba con 98.60%, seguido de Ipomoea nil con 97.73% y la secuencia que presentó menor porcentaje de similitud de las secuencias seleccionadas fue Carica papaya con 80.90% (Tabla 1). La cercanía de las secuencias de aminoácidos del gen fitoeno desaturasa en camote con Ipomoea triloba e Ipomoea nil se evidencia también en el árbol filogenético (Figura 2) y presentan similitud entre las tres especies con la presencia de gaps entre el aminoácido 15 y 23, en comparación con las otras secuencias que no presentan dicha característica (Figura 3). Este alto porcentaje de similitud entre las secuencias se atribuye a la divergencia evolutiva que presenta el camote con Ipomoea triloba e Ipomoea nil, específicamente este último presenta una divergencia del linaje que contiene con Ipomoea triloba, de esta manera considerando a Ipomoea nil como el posible ancestro común entre ambas especies18.
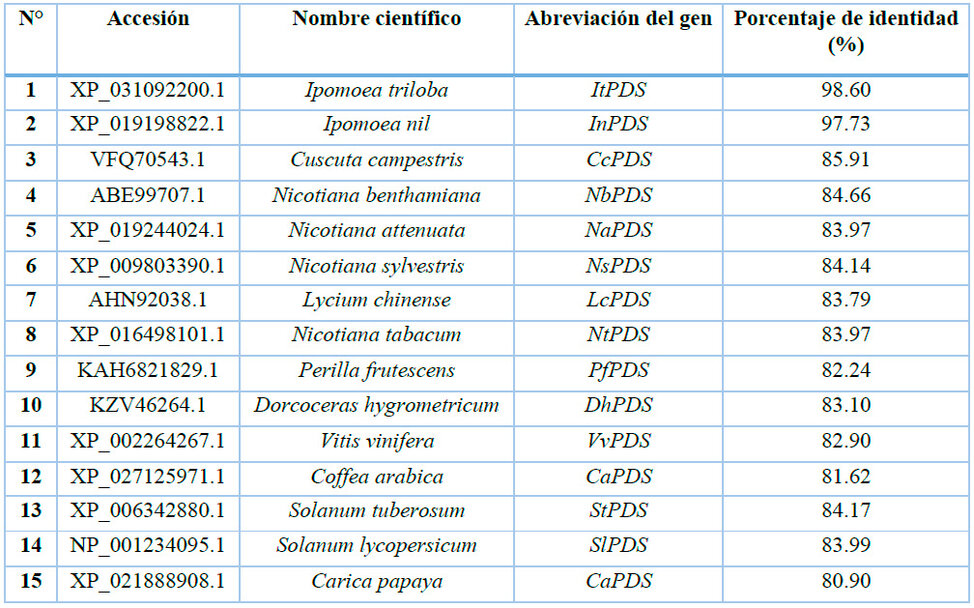
Tabla 1: Accesiones del gen PDS de especies vegetales seleccionadas para alineamiento múltiple de secuencias de aminoácidos para el gen IbPDS.
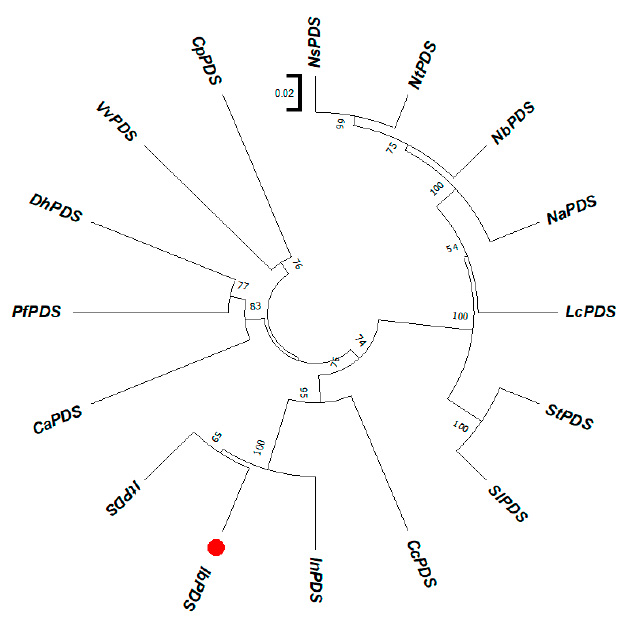
Figura 2: Análisis de árbol filogenético de unión de vecinos de la proteína PDS de camote y especies cercanas.

Figura 3: Comparación de las secuencias de aminoácidos de PDS de camote y especies cercanas.
Diseño in silico de sgRNA para el gen IbPDS
En el diseño in silico de los sgRNA se identificaron 113 secuencias candidatas para la edición genética en la región del gen IbPDS. Mediante el filtrado de las secuencias se redujo a un total de 24 sgRNA o RGENs candidatos (Tabla 2), el filtrado aplicado estuvo constituido por la evaluación en 0 para Desajustes 1 y posterior se agregaron filtros adicionales en 0 para los Desajustes 1 y 2 en número de objetivos encontrados, permitiendo la reducción hasta de 19 sgRNA secuencias final.
Las consideraciones para la aplicación de filtros estuvieron basadas en Malik et al.19, quienes mencionan que la eficacia de la escisión sgRNA se ve influida positivamente por el aumento del contenido de guanina (G) y citosina (C), pero no obstante el aumento del contenido GC disminuye las actividades de escisión notablemente. La elección de los valores de puntuación fuera de cuadro es importante elegir sitios de alta puntuación porque aumenta la probabilidad de obtener clones mutantes permanentes. Establecemos que el contenido de GC para este trabajo debe presentar un rango de 40-60% porque son valores eficientes demostrado por Bae et al.20, siendo el primer filtro y seguido del segundo filtro mediante la aplicación discriminante de puntuación fuera cuadro de edición genómica, el cual debe presentar un valor superior a 66 como recomienda la herramienta en línea CRISPR RGEN Tools.

*Secuencias de color azul fueron las secuencias de sgRNA seleccionadas.
Tabla 2: Secuencias de sgRNA para la edición del genómica en la región del gen IbPDS.

Tabla 3: Secuencias objetivo/fuera de objetivo para edición del genoma de región del gen IbPDS.
Los 19 sgRNA seleccionadas de la (Tabla 1) proporcionaron mediante la función Cas-OFFinder 50 secuencias objetivas (Tabla 2) con 323 sitios incisión probables en el genoma entre sitios objetivos y sitios fuera del objetivo que difieren en varios nucleótidos de los sitios en el objetivo. Mediante el filtro de las secuencias objetivos se redujo a 25 secuencias (Tabla 2), el filtro aplicado se considera las secuencias objetivo que pueden poseer “1” en número de objetivos encontrados. Además, se identificaron las 19 secuencias objetivo-finales que son equivalente a 19 sgRNA (Tabla 3), ya que poseen solo un sitio objetivo en el gen IbPDS ubicado en el cromosoma 8 y no poseen sitios fuera del objetivo. Cho et al.21, mencionan que las RGEN pueden distinguir los sitios de interés de los que no lo son con desajustes de dos bases, pero no aquellos con un desajuste de una sola base. En su investigación trabajó con células K562 y demostró que dos RGENs indujeron indels en frecuencias del 75% (C4BPB) y del 60% (CCR5) en los sitios correspondientes en las células K562, además los RGEN no pueden escindir eficientemente al DNA cromosómico si poseen desajustes de dos o más nucleótidos. Por otra parte, RGEN pueden distinguir los sitios objetivo de los sitios fuera del objetivo que difieren en al menos dos bases, permitiendo elegir sitios objetivo-únicos que no tengan secuencias homólogas en otras partes del genoma.
Predicción in silico de la estructura de sgRNA para IbPDS
Las estructuras secundarias de los tres sgRNA seleccionados fueron predichas presentando la formación de dos horquillas en el extremo superior y una doble horquilla en el extremo del tallo inferior (Figura 4). La determinación de estas estructuras secundarias es importante porque permite identificar las secuencias de sgRNA que pueden presentar variaciones estructurales que mermen la capacidad de reconocer y editar la región diana22,23. Sin embargo, dentro de estas potenciales variaciones, se establece la presencia de un motivo conservado en el sgRNA, que determina la formación de estructuras en forma de horquillas estables en posiciones establecidas, permitiendo la obtención de estructuras que están en concordancia con ser reconocido como un componente clave en el diseño de sgRNAs24.
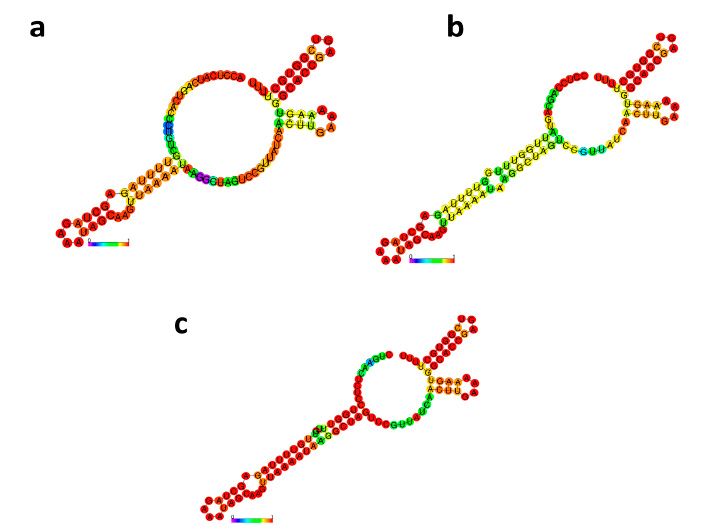
Figura 4: Estructuras secundarias de los sgRNA para la edición genética del gen IbPDS: (a) sgRNA 1, (b) sgRNA 2 y (c) sgRNA 3.
Diseño in silico del vector de inactivación del gen IbPDS mediada por CRISPR/Cas9
Se diseñó el vector de destino pMH-Cas9-3xsgRNA para la edición genética del gen IbPDS, la inserción de los tres sgRNA se realizó in silico mediante las reacciones de recombinación direccional mulisitio Gateway LR, específicamente en los sitios att1 y att2 del vector inicial. El vector presenta, a parte de los tres sgRNA, el marcador de selección de resistencia a Higromicina, el cual cuenta con el promotor 35S; también está presente la secuencia de la endonucleasa Cas9 con su promotor Ubi y terminador 35S (Figura 5). El vector de destino fue diseñado para la transformación genética de protoplastos de camote, según la metodología establecida por Nishimaki y Nozué25, para la obtención de los protoplastos de camote y modificada para la aplicación del vector según la metodología establecida por Steiner26, quienes establecieron metodologías para un alto rendimiento de obtención de protoplastos y la regeneración de pequeñas colonias a partir de los protoplastos hasta obtener plantas completas, así como una eficiencia de transformación cercano al 90%.
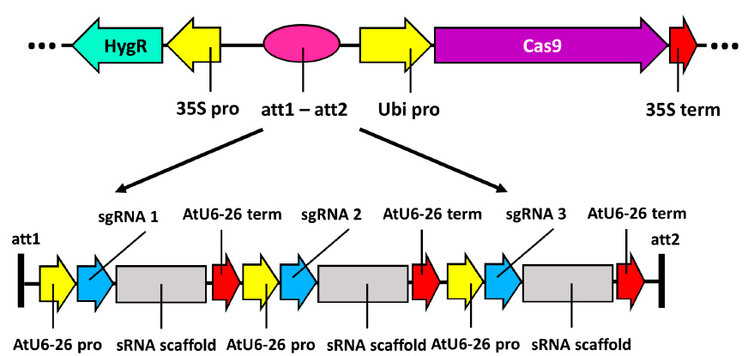
Figura 5: Mapa del vector de destino pMH-Cas9-3xsgRNA para la edición genética del gen IbPDS.
El diseño de vectores in silico, es una herramienta que permite predecir con precisión los sitios de restricción necesarios para la inserción de la máquina de edición genética, como es el caso de los tres sgRNA con sus respectivos promotores y terminadores. En otras investigaciones también utilizan SnapGene como herramienta para la construcción de un vector de expresión insertado con la secuencia de sgRNA pero en este caso emplearon un promotor T7 y otros promotores virales que codifican tanto el sgRNA como la proteína Cas1227. Este tipo de herramientas computacionales permite manipular las secuencias que codifican genes para la proteína Cas como para las resistencias a antibióticos, lo que representa una ventaja a tener en cuenta para las futuras etapas experimentales en laboratorio, evitando de esta manera futuros errores y por lo tanto reduciendo o evitando también perdidas en el uso de reactivos y tiempo por posibles fallos durante el clonamiento de este tipo de vectores28. Las herramientas del diseño de vectores in silico permiten además seleccionar y manipular diversos tipos de vectores, dependiendo del organismo a trabajar, así se presentan metodos efectivos para diseñar vectores de expresión transitoria o por métodos Gateway29.
El uso de las herramientas computacionales o in silico son cruciales para prever la precisión y el éxito de la edición génica mediada por CRISPR/Cas9 del gen IbPDS en camote. Todas las herramientas utilizadas presentan el enfoque computacional, gracias a los avances y mejoras de algoritmos, el diseño de los sgRNA permite predecir la varianza individual relacionada con las actividades dentro y fuera del objetivo30. De esta manera los métodos in silico cubren las características más específicas, como el entorno de la cromatina, la accesibilidad y la expresión de exones, y ofrecen predicciones más precisas31. Considerando también la combinación del predictor de microhomología y la alineación local con la referencia puede allanar un nuevo camino para cuantificar las mutaciones inducidas en la secuencia del gen IbPDS32. El diseño in silico de sgRNA proporciona un paso clave para el sistema CRISPR y permite que los estudios CRISPR aprovechen la bioinformática y las técnicas computacionales, precisamente mediante el uso de estas herramientas computacionales se permite el diseño de sgRNA in silico con una alta eficacia en el objetivo y una reducción de los efectos fuera del objetivo33.
CONCLUSIONES
Se diseñó in silico tres sgRNA para la inactivación del gen IbPDS mediada por CRISPR/Cas9. Se comparó la secuencia de aminoácidos de PDS de camote con otras especies vegetales y se demostró que presentan una similitud cercana con PDS de Ipomoea triloba e Ipomoea nil con 98.60% y 97.73%, respectivamente. La predicción de las estructuras secundarias de los sgRNA seleccionados presentan estructuras de sgRNA eficientes en edición genética. Así mismo, se diseñó el vector pMH-Cas9-3xsgRNA, vector con las tres secuencias de sgRNA diseñadas. Este vector de edición genética fue diseñado mediante herramientas computacionales in silico, al igual que los sgRNA y las predicciones de estructuras secundarias. Estas herramientas son importantes en la predicción de la precisión y éxito de la edición genética del gen objetivo, mediante este enfoque permite a los estudios moleculares enfocados a edición genética aprovechar la bioinformática y técnicas computacionales para diseñar y predecir de manera práctica moléculas de ADN o proteínas.
Contribuciones de los autores: Conceptualización, MYC y BL; metodología, MYC, BL, GAB y ALG; software, MAALV y EDR; validación, DECD, CRPR, LRV; análisis formal, DECD, CRPR, MAALV y LRV; investigación, MYC, BL, GAB y ALG; conservación de datos, MYC y BL; redacción del borrador original, MYC, BL, GAB y ALG; redacción, revisión y edición, DECD, CRPR, EDR, MAALV y LRV; supervisión, LRV; Todos los autores han leído y aceptado la versión publicada del manuscrito.
Conflictos de intereses: Los autores declaran no tener ningún conflicto de intereses.
REFERENCIAS
1. Mojica, F.J.; Montoliu, L. On the Origin of CRISPR-Cas Technology: From Prokaryotes to Mammals. Trends in Microbiology 2016, 24, 811-820.
2. Wang, H.; La-Russa, M.; Qi, L.S. CRISPR/Cas9 in Genome Editing and Beyond. Annual Reviews in Biochemistry 2016, 85, 227-64.
3. Jain, A.; Zode, G.; Kasetti, R.B.; Ran, F.A.; Yan, W.; Sharma, T.P.; Bugge, K.; Seaby, C.C.; Fingert, J.H.; Zhang, F.; Clark, A.; Sheffield, A.C. CRISPR-Cas9–based treatment of myocilin-associated glaucoma. Proc Natl Acad Sci USA 2017,114(42), 11199-11204.
4. Komor, A.; Kim, Y.; Packer, M.; Zuris, J.A.; Liu, D.R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 2016, 533, 420-424.
5. Jaganathan, D.; Ramasamy, K.; Sellamuthu, G.; Jayabalan, S.; Venkataraman, G. CRISPR for Crop Improvement: An Update Review. Front. Plant Sci. 2018, 9, 985.
6. Doudna, J.A.; Charpentier, E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science 2014, 346, 1258096.
7. Oke, M.; Workneh, T.S. A review on sweet potato postharvest processing and preservation technology. African Journal of Agricultural Research 2013, 8(40), 4990-5003.
8. James, M.G.; Robertson, D.S.; Myers, A.M. Characterization of the maize gene sugary1, a determinant of starch composition in kernels. Plant Cell. 1995, 7(4), 417-29.
9. Seo, S.G.; Jang, H.R.; Shin, J.M.; Jun, B.K.; Shim, I-S.; Kim, S-H. Ectopic expression of ibPDS gene enhanced tolerance to oxidative stress in transgenic tobacco plants. Plant Growth Regul 2015, 77, 245-253.
10. Wang, H.; Wu, Y.; Zhang, Y.; Yang, J.; Fan, W.; Zhang, H.; Zhao, S.; Yuan, L.; Zhang, P. CRISPR/Cas9-Based Mutagenesis of Starch Biosynthetic Genes in Sweet Potato (Ipomoea Batatas) for the Improvement of Starch Quality. Int J Mol Sci. 2019, 20(19), 4702.
11. Hernández-Amasifuen, A.D.; Cortez-Lázaro, A.A.; Argüelles-Curaca, A.; Díaz-Pillasca, H.B. In vitro callogenesis of peach (Prunus persica L.) var. Huayco rojo from leaf explants. Ciencia Tecnología Agropecuaria 2021, 23(1), e2032.
12. Hernández-Amasifuen, A.D.; Argüelles-Curaca, A.; Cortez-Lázaro, A.A.; Díaz-Pillasca, H.B. In vitro induction of callus from foliar explants in rocoto (Capsicum pubescens Ruiz & Pav.). Granja 2021, 34(2), 127-135.
13. Hernández-Amasifuen, A.D.; Pineda-Lázaro, A.J.; Díaz-Pillasca, H.B. In vitro anther culture of rocoto (Capsicum pubescens Ruiz & Pav.). Idesia 2022, 40(1), 115-121.
14. Pineda-Lázaro, A.J.; Hernández-Amasifuen, A.D.; Díaz-Pillasca, H.B. The efficient procedure of embryogenic callus formation from anther in Capsicum pubescens Ruiz & Pav. Bionatura 2023, 8(1), 29.
15. Hernández-Amasifuen, A.D.; Pineda-Lázaro, A.J.; Díaz-Pillasca, H.B. In vitro micropropagation of sour orange (Citrus aurantium L.) from nodal segments. Bionatura 2021, 6(4), 2216-2221.
16. Gao, X.; Chen, J.; Dai, X.; Zhang, D.; Zhao, Y. An Effective Strategy for Reliably Isolating Heritable and Cas9-Free Arabidopsis Mutants Generated by CRISPR/Cas9-Mediated Genome Editing. Plant Physiol. 2016, 171(3), 1794-1800.
17. Mallett, D.R.; Chang, M.; Cheng, X.; Bezanilla, M. Efficient and modular CRISPR-Cas9 vector system for Physcomitrella patens. Plant Direct. 2019, 3, 1-15.
18. Wu, S.; Lau, K.H.; Cao, Q.; et al. Genome sequences of two diploid wild relatives of cultivated sweetpotato reveal targets for genetic improvement. Nat Commun 2018, 9, 4580.
19. Malik, A.; Gul, A.; Munir, F.; Amir, R.; Alipour, H.; Babar, M.M.; Bakhtiar, S.M.; Paracha, R.Z.; Khalid, Z.; Hayat, M.Q. Evaluating the cleavage efficacy of CRISPR-Cas9 sgRNAs targeting ineffective regions of Arabidopsis thaliana genome. PeerJ 2021, 9, e11409.
20. Bae, S.; Kweon, J.; Kim, H. et al. Microhomology-based choice of Cas9 nuclease target sites. Nat Methods 2014, 11, 705-706.
21. Cho, S.W.; Kim, S.; Kim, Y.; Kweon, J.; Kim, H.S.; Bae, S.; Kim, J-S. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 2014, 24, 132-141
22. Robins, H.; Li, Y.; Padgett, R.W. Incorporating structure to predict microRNA targets. Proc Natl Acad Sci USA 2005, 102(11), 4006-4009.
23. Konstantakos, V.; Nentidis, A.; Krithara, A.; Paliouras, G. CRISPR-Cas9 gRNA efficiency prediction: an overview of predictive tools and the role of deep learning. Nucleic Acids Res 2022, 50(7), 3616-3637.
24. Wong, N.; Liu, W.; Wang, X. WU-CRISPR: characteristics of functional guide RNAs for the CRISPR/Cas9 system. Genome Biol 2015, 16(1), 218.
25. Nishimaki, T.; Nozué, M. Isolation and culture of protoplasts from high anthocyanin-producing callus of sweet potato. Plant Cell Rep 1985, 4(5), 248-251.
26. Steiner, J. Protoplast isolation and transformation in sweetpotato. Faculty of Engineering, Lund University, Lund, Suecia, 2022.
27. Karlapudi, A.; Thirupati, C.; Srirrama, K.; Nageswaran, D.; Mikkili, I.; Ayyagari V. Design of CRISPR-Based Targets for the Development of a Diagnostic Method for SARS-CoV-2: An in Silico Approach. EJMO 2020, 4(4), 304-308.
28. Hernández-Amasifuen, A.D.; Yupanqui-Celestino, M.; Pineda-Lázaro, A.J.; Delgado-Mera, E.; Ramírez-Viena, L.; Pesantes-Rojas, C.R.; Corazon-Guivin, M.A. In silico design of sgRNA for CRISPR/Cas9-mediated FaRALF33 gene mutagenesis to decrease the infection process to Colletotrichum acutatum in strawberry. J App Biol Biotech 2024, 12(3), 1-8.
29. Alburqueque-Vasquez, N.J.; Hernández-Amasifuen, A.D.; Pineda-Lázaro, A.J.; Fernández Ponce, J.N.; Guerrero-Abad, J.C.; García, L.; Corazon-Guivin, M.A. Response of the transcription factor BABY BOOM of Arabidopsis thaliana L. in the formation of embryogenic calluses of cocoa leaves (Theobroma cacao L.). Scientia Agropecuaria 2023, 14(3), 359-366.
30. Canver, M.C.; Lessard, S.; Pinello, L.; Wu, Y.; Ilboudo, Y.; Stern, E.N. Variant-aware saturating mutagenesis using multiple Cas9 nucleases identifies regulatory elements at trait-associated loci. Nat Genet 2017, 49, 625-34.
31. Lessard, S.; Francioli, L.; Alfoldi, J.; Tardif, J.C.; Ellinor, P.T.; Macarthur, D.G. Human genetic variation alters CRISPR-Cas9 on- and off-targeting specificity at therapeutically implicated loci. Proc Natl Acad Sci USA 2017, 114, E11257-66.
32. Liu, G.; Yin, K.; Zhang, Q.; Gao, C.; Qiu, J.L. Modulating chromatin accessibility by transactivation and targeting proximal dsgRNAs enhances Cas9 editing efficiency in vivo. Genome Biol 2019, 20, 145.
33. Zhang, Y.; Zhao, G.; Ahmed, F.Y.H.; Yi, T.; Hu, S.; Cai, T.; Liao, Q. In silico Method in CRISPR/Cas System: An Expedite and Powerful Booster. Front Oncol 2020, 10, 584404.
Received: October 9th 2023/ Accepted: January 15th 2024 / Published:15 February 2024
Citation: Yupanqui-Celestino M., Lugo B., Aguilar-Balabarca G. J., Lozano-Galindo A. E., Cotos-Durán D. E., Pesantes-Rojas C. R., Aguilar-Luna-Victoria M. A., Díaz-Ronceros E., Ramírez-Viena, L. In silico design of CRISPR/Cas9 guide RNA for the knockout of the phytoene desaturase gene in sweet potato (Ipomoea batatas L.). Revis Bionatura 2024; 1 (1) 74. http://dx.doi.org/10.21931/BJ/2024.01.01.74
Additional information Correspondence should be addressed to [email protected]
Peer review information. Bionatura thanks anonymous reviewer(s) for their contribution to the peer review of this work using https://reviewerlocator.webofscience.com/
All articles published by Bionatura Journal are made freely and permanently accessible online immediately upon publication, without subscription charges or registration barriers.
Publisher's Note: Bionatura stays neutral concerning jurisdictional claims in published maps and institutional affiliations.
Copyright: © 2024 by the authors. They were submitted for possible open-access publication under the terms and conditions of the Creative Commons Attribution (CC BY) license (https://creativecommons.org/licenses/by/4.0/).
